Vírus
| Vírus | |||||
|---|---|---|---|---|---|
| Classificação científica | |||||
| |||||
| Grupos | |||||
| |||||
| Sinónimos | |||||
| |||||
Vírus (do latim virus, "veneno" ou "toxina") são pequenos agentes infecciosos, a maioria com 20-300 nm de diâmetro, apesar de existirem vírus ɡiɡantes de (0,6–1,5 µm), que apresentam genoma constituído de uma ou várias moléculas de ácido nucleico (DNA ou RNA), as quais possuem a forma de fita simples ou dupla. Os ácidos nucleicos dos vírus geralmente apresentam-se revestidos por um envoltório proteico formado por uma ou várias proteínas, que pode ainda ser revestido por um complexo envelope formado por uma bicamada lipídica.[1][2]
As partículas virais são estruturas extremamente pequenas, submicroscópicas. A maioria dos vírus é tão diminuta que está além dos limites de resolução dos microscópios ópticos comuns. Por isso, para a sua visualização é frequente o uso de microscópios eletrônicos. Vírus são estruturas simples, se comparados a células, e não são considerados organismos, pois não possuem organelas ou ribossomos, e não apresentam todo o potencial bioquímico (enzimas) necessário à produção de sua própria energia metabólica. Eles são considerados parasitas intracelulares obrigatórios (característica que os impede de serem considerados seres vivos), pois dependem de células para se multiplicarem. Além disso, diferentemente dos organismos vivos, os vírus são incapazes de crescer em tamanho e de se dividir. A partir das células hospedeiras, os vírus obtêm: aminoácidos e nucleotídeos; maquinaria de síntese de proteínas (ribossomos) e energia metabólica (ATP).[3][4][5]
Fora do ambiente intracelular, os vírus são inertes.[1][2] Porém, uma vez dentro da célula, a capacidade de replicação dos vírus é surpreendente: um único vírus é capaz de multiplicar, em poucas horas, milhares de novos vírus. Os vírus são capazes de infectar seres vivos de todos os domínios (Eukarya, Archaea e Bactéria). Desta maneira, os vírus representam a maior diversidade biológica do planeta, sendo mais diversos que bactérias, plantas, fungos e animais juntos.[4][5] Quase 200 mil tipos diferentes de vírus se espalham nos oceanos do mundo, de acordo com um estudo. A contagem de 2019 é 12 vezes maior do que o censo anterior de vírus marinhos registrado em 2016.[6]
Existem, individualmente, cerca de dez nonilhões (10³¹) de vírus no planeta Terra, uma quantidade cem milhões de vezes maior que o número de estrelas no universo observável.[7]
Etimologia
[editar | editar código-fonte]A palavra "vírus" vem do latim virus, que se refere a veneno, ou alguma substância nociva. Foi usada pela primeira vez, em inglês, em 1392. O termo "virulento", do latim virulentus (venenoso), data de 1400.[8][9] O termo "virion" também é usado para se referir a uma única partícula viral infecciosa.[10]
Histórico
[editar | editar código-fonte]
Em meados do século XIX, Louis Pasteur propôs a teoria microbiana das doenças, na qual explicava que todas as doenças eram causadas e propagadas por algum “tipo de vida diminuta” que se multiplicava no organismo doente, se transmitia para outro e o contaminava. Pasteur, no entanto, ao trabalhar com a raiva, constatou que, embora a doença fosse contagiosa e transmitida pela mordida de um animal raivoso, o micro-organismo não podia ser observado. Pasteur concluiu que o agente infeccioso estava presente mas era muito pequeno para ser observado através do microscópio.[11]
Em 1884, o microbiologista Charles Chamberland desenvolveu um filtro (conhecido como filtro Chamberland ou Chamberland-Pasteur), com poros mais pequenos do que uma bactéria. Fazendo passar uma solução que continha bactérias através desse filtro, as bactérias ficavam nele retidas e a solução filtrada obtida tornava-se estéril.[12] Em 1886, Adolf Mayer demonstrou que a doença do tabaco podia ser transmitida a plantas saudáveis pela inoculação com extratos de plantas doentes.[13][14] Em 1892, o biólogo Dmitry Ivanovsky fez uso do filtro Chamberland para demonstrar que folhas de tabaco infectadas trituradas continuavam infectadas mesmo após a filtragem.[15] Ivanovsky sugeriu que a infecção poderia ser causada por uma toxina produzida pelas bactérias, mas ele não persistiu nesta hipótese. Em 1898, o microbiologista Martinus Beijerinck repetiu a experiência independentemente e ficou convencido que a solução filtrada continha um novo agente infeccioso, denominado de contagium vivum fluidum (fluido vivo contagioso).[16] Ele também observou que este agente apenas se reproduzia em células que se dividiam, mas não conseguiu determinar se este seria constituído de partículas, assumindo que os vírus estariam presentes no estado líquido.[17] Beijerinck introduziu o termo 'vírus' para indicar que o agente causal da doença do mosaico do tabaco não tinha uma natureza bacteriana, e sua descoberta é considerada como o marco inicial da virologia. A teoria do estado líquido do agente foi questionada nos 25 anos seguintes, sendo descartada com o desenvolvimento de teste da placa por d'Herelle em 1917,[18][19] pela cristalização desenvolvida por Wendell Meredith Stanley em 1935[17][20] e pela primeira microfotografia eletrônica realizada em 1939 do vírus do mosaico do tabaco.[21]
Em 1898, Friedrich Loeffler e Paul Frosch identificaram o primeiro agente filtrável de animais, o vírus da febre aftosa (Aphtovirus). E em 1901, Walter Reed identificou o primeiro vírus humano, o vírus da febre amarela (Flavivirus).[22] Em 1908, Vilhelm Ellerman e Olaf Bang demonstraram o potencial oncogênico de um agente filtrável, descobrindo o vírus da leucose aviária.[23] E em 1911, Peyton Rous transmitiu um tumor maligno de uma galinha para outra, descobrindo o vírus do sarcoma de Rous, e demonstrando que o câncer poderia ser transmitido por um vírus.[24]
Em 1915, o bacteriologista Frederick William Twort ao tentar propagar o vírus da vaccínia num meio de cultura bacteriana observou que as colônias morriam e que o agente dessa transformação era infeccioso. Twort propôs várias explicações para o ocorrido, como uma ameba, um protoplasma, um vírus ultramicroscópico ou uma enzima que afetava o crescimento.[25][26] Independentemente, em 1917, o microbiologista Félix Hubert d'Herelle descobriu que colônias bacterianas eram atacadas por um agente e imediatamente o reconheceu como sendo um vírus, cunhando o termo bacteriófago. Ele utilizou os fagos para o tratamento de doenças bacterianas e fundou diversos institutos de fagos em vários países.[25][19]
Inicialmente, o único meio para recuperar quantidades significativas de vírus era por meio de infecção em animais suscetíveis.[27] Em 1913, Edna Steinhardt e colaboradores conseguiram fazer crescer o vírus da vaccínia em fragmentos de córneas de cobaias.[28] Em 1928, H.B. Maitland e M.C. Maitland cultivaram o vírus de vaccínia em suspensão de rins de galinhas moídos.[29] Em 1931, o patologista Ernest William Goodpasture cultivou o vírus da varíola aviária na membrana corioalantóide de ovos de galinhas embrionados.[30] Em 1937, Max Theiler cultivou o vírus da febre amarela em ovos de galinha e desenvolveu uma vacina a partir de uma estirpe do vírus atenuado.[31] Em 1949, John Franklin Enders, Thomas Weller e Frederick Robbins cultivaram o vírus da poliomielite em culturas de células embrionárias humanas, o primeiro vírus a ser cultivado sem a utilização de tecido animal sólido ou ovos.[32] Este método permitiu a Jonas Salk desenvolver uma vacina eficaz contra a poliomielite.[33]
As primeiras imagens de vírus foram obtidas após a invenção do microscópio eletrônico em 1931 pelos engenheiros Ernst Ruska e Max Knoll. Em 1935, o bioquímico e virologista Wendell Meredith Stanley examinou o vírus do mosaico do tabaco e descobriu que o mesmo era constituído principalmente por proteínas.[34] Em 1937, Frederick Bawden e Norman Pirie separaram o vírus do mosaico em porções proteicas e de RNA.[35] O vírus do mosaico do tabaco foi o primeiro a ser cristalizado e, por conseguinte, a sua estrutura pode ser analisada em detalhes. As primeiras imagens de raios-X de difração do vírus cristalizado foram obtidas por Bernal e Fankuchen em 1941.[36] Com base nos seus quadros, Rosalind Franklin descobriu a estrutura completa do vírus em 1955.[37] No mesmo ano, Heinz Fraenkel-Conrat e Robley Williams demonstraram que o RNA do vírus do mosaico do tabaco e o seu revestimento de proteína purificada (capsídeo) podiam montar-se por si só para formar vírus funcionais, sugerindo que este mecanismo simples foi, provavelmente, o meio pelo qual os vírus foram replicados dentro das células hospedeiras.[1]
A segunda metade do século XX foi a idade de ouro da descoberta do vírus e foram reconhecidas mais de 2 000 novas espécies de vírus de animais, plantas e bactérias.[38] Em 1957, descobriu-se o arterivírus equino e o vírus da diarreia bovina (um pestivírus). Em 1963, Baruch Blumberg descobriu o vírus da hepatite B, e em 1965, Howard Temin descreveu o primeiro retrovírus. A transcriptase reversa, que é a enzima fundamental dos retrovírus, que utilizam para copiar o seu ARN para ADN, foi descrita em 1970, por Howard Martin Temin e David Baltimore, de forma independente.[39] Em 1983, a equipe de Luc Montagnier do Instituto Pasteur, na França, isolou pela primeira vez o retrovírus que hoje conhecemos por HIV.[40]
Taxonomia
[editar | editar código-fonte]Classificação taxonômica
[editar | editar código-fonte]Os vírus também são classificados dentro de grupos taxonômicos, assim como os seres vivos, porém, seguindo uma regra particular de classificação. Vírus não são agrupados em domínio, reino, filos ou classes. Desta maneira, a estrutura geral da taxonomia dos vírus é a seguinte:[5]
- Ordem (-virales);
- Família (-viridae);
- Subfamília (-virinae);
- Gênero (-virus);
- Espécie.
A nomenclatura para ordens, famílias, subfamílias e gêneros é sempre precedida pelos sufixos apresentados acima. Já a nomenclatura de espécies não possui um padrão universal. Cada ramo da virologia (vegetal, animal, bacteriana) adota um padrão de nomenclatura específico. Espécies de vírus de plantas normalmente apresentam nomes que fazem referência a planta hospedeira e a característica do sintoma causado pela infecção (e.g. Vírus do mosaico do tabaco). Espécies de vírus de bactérias (bacteriófagos) podem ser denominados como "fago" seguido de uma letra grega (e.g. Fago λ) ou código alfanumérico (e.g. Fago T7). Vírus que infectam vertebrados podem receber nomes em alusão à espécie hospedeira de origem (e.g. Papillomavírus Bovino), ao local de origem do vírus (e.g. Vírus Ebola, do rio Ébola, no Congo), à doença causada pelo vírus (e.g. Vírus da imunodeficiência humana - HIV).[41]
O Comitê Internacional de Taxonomia de Vírus (ICTV, do inglês "International Committee on Taxonomy of Virus") estabelece regras de classificação e nomenclatura de vírus. O ICTV é uma entidade composta por grupos especializados de virologistas de todas as partes do mundo.[5]

Classificação de Baltimore
[editar | editar código-fonte]O Sistema de Classificação de Baltimore, criado por David Baltimore, é um modo de classificação que ordena os vírus em sete grupos, com base na característica do genoma viral e na forma como este é transcrito a mRNA. Neste sistema, os vírus são agrupados como apresentado a seguir:[5]
- Grupo I: Vírus DNA dupla fita (dsDNA);
- Grupo II: Vírus DNA fita simples (ssDNA);
- Grupo III: Vírus RNA dupla fita (dsRNA);
- Grupo IV: Vírus RNA fita simples senso positivo ((+)ssRNA);
- Grupo V: Vírus RNA fita simples senso negativo ((-)ssRNA);
- Grupo VI: Vírus RNA com transcrição reversa (ssRNA-RT);
- Grupo VII: Vírus DNA com transcrição reversa (dsDNA-RT).
Vírus de DNA
[editar | editar código-fonte]No vírus de DNA, a replicação do genoma da maioria dos vírus de DNA ocorre no núcleo da célula. Se a célula possui o receptor apropriado na superfície, esses vírus entram por fusão com a membrana celular ou por endocitose. A maioria dos vírus de DNA é completamente dependente das máquinas de síntese de DNA e RNA da célula hospedeira e de suas máquinas de processamento de RNA. O genoma viral deve passar pelo envelope nuclear da célula para acessar esse mecanismo.[42][43]
Vírus de RNA
[editar | editar código-fonte]Os vírus de RNA são únicos porque suas informações genéticas são codificadas em RNA; Isso significa que eles usam ácido ribonucleico (RNA) como material genético ou que, no processo de replicação, precisam de RNA. A replicação geralmente ocorre no citoplasma.[44] Os vírus de RNA podem ser classificados em cerca de quatro grupos de acordo com seu modo de replicação. A polaridade do RNA (se pode ou não ser usado diretamente para produzir proteínas) determina em grande parte o mecanismo de replicação e se o material genético é de fita simples ou dupla. Os vírus de RNA usam suas próprias replicasses de RNA para criar cópias de seu genoma.[45][46]
A origem
[editar | editar código-fonte]A origem dos vírus não é totalmente clara, e provavelmente, esta seja tão complexa quanto a origem da vida. Porém, foram propostas algumas hipóteses.[1] Além disso, o material genético viral ocasionalmente se integra à linha germinativa dos organismos hospedeiros, pela qual eles podem ser passados verticalmente para os filhotes do hospedeiro por muitas gerações.[47] Isso fornece uma fonte inestimável de informações para os paleovirologia rastrearem vírus antigos que existiam há milhões de anos. Existem três hipóteses principais que visam explicar as origens dos vírus:[48]
- Hipótese regressiva: Os vírus podem ter sido células pequenas que parasitaram células maiores. Com o tempo, os genes não exigidos pelo parasitismo foram perdidos. As bactérias rickettsia e clamídia são células vivas que, como os vírus, podem se reproduzir apenas dentro das células hospedeiras.[49] Eles apoiam essa hipótese, pois sua dependência do parasitismo provavelmente causou a perda de genes que lhes permitiram sobreviver fora de uma célula. Isso também é chamado de 'hipótese da degeneração', ou 'hipótese de redução';[50]
- Hipótese de origem celular: Alguns vírus podem ter evoluído a partir de pedaços de DNA ou RNA que "escaparam" dos genes de um organismo maior. O DNA escapado pode ter vindo de plasmídeos (pedaços de DNA nu que podem se mover entre células) ou transposons (moléculas de DNA que se replicam e se movem para diferentes posições dentro dos genes da célula).[51] Uma vez chamados de "genes saltadores", os transposons são exemplos de elementos genéticos móveis e podem ser a origem de alguns vírus. Eles foram descobertos no milho por Barbara McClintock em 1950.[52] Às vezes, isso é chamado de "hipótese da vaga",ou "hipótese da fuga";[50]
- Hipótese de co-evolução: Isso também é chamado de "hipótese do vírus primeiro"[50] e propõe que os vírus podem ter evoluído a partir de moléculas complexas de proteínas e ácidos nucleicos ao mesmo tempo em que as células apareceram pela primeira vez na Terra e que seriam dependentes da vida celular por bilhões de anos. Os viróides são moléculas de RNA que não são classificadas como vírus porque não possuem uma camada protéica. Eles têm características comuns a vários vírus e são frequentemente chamados de agentes subvirais. Os viróides são patógenos importantes das plantas. Eles não codificam proteínas, mas interagem com a célula hospedeira e usam o mecanismo hospedeiro para sua replicação.[53] O vírus da hepatite delta em humanos possui um genoma de RNA semelhante aos viróides, mas possui uma camada protéica derivada do vírus da hepatite B e não pode produzir um deles. É, portanto, um vírus defeituoso. Embora o genoma do vírus da hepatite delta possa se replicar independentemente uma vez dentro de uma célula hospedeira, ele requer a ajuda do vírus da hepatite B para fornecer um revestimento protéico para que possa ser transmitido para novas células.[49]
No passado, havia problemas com todas essas hipóteses: a hipótese regressiva não explicava por que mesmo os menores parasitas celulares não se assemelham a vírus de forma alguma. A hipótese de escape não explicou os capsídeos complexos e outras estruturas nas partículas virais. A hipótese do primeiro vírus violou a definição de vírus, na medida em que requerem células hospedeiras.[50] Os vírus agora são reconhecidos como antigos e como tendo origens anteriores à divergência da vida nos três domínios. Essa descoberta levou os virologistas modernos a reconsiderar e reavaliar essas três hipóteses clássicas.[54]
Genoma
[editar | editar código-fonte]| Propriedade | Parâmetros |
|---|---|
| Ácido nucleico |
|
| Forma |
|
| Estrutura |
|
| Sentido |
|
Ao contrário das células, que apresentam genoma constituído por DNA e RNA, os vírus possuem DNA ou RNA como material genético, e todos os vírus possuem apenas um ou outro no vírion. No entanto, existem vírus que possuem ambos, porém, em estágio diferentes do ciclo reprodutivo.[55] As moléculas de ácido nucleico dos vírus podem ser fita simples ou dupla, linear ou circular, e segmentada ou não. O genoma dos vírus de RNA tem ainda a característica de possuir senso positivo (atua como mRNA funcional no interior das células infectadas) ou senso negativo (serve de molde para uma RNA-polimerase transcrevê-lo dando origem a um mRNA funcional).[2] A quantidade de material genético viral é menor que a da maioria das células.[55] O peso molecular do genoma dos vírus de DNA varia de 1,5 × 106 a 200 × 106 Da. Já o dos de RNA varia de 2 × 106 a 15 × 106 Da. No genoma dos vírus estão contidas todas as informações genéticas necessárias para programar as células hospedeiras, induzindo-as a sintetizar todas as macromoléculas essenciais à replicação do vírus.[2]
Um genoma viral, independentemente do tipo de ácido nucleico, é quase sempre de fita simples ou dupla. Os genomas de fita simples consistem em um ácido nucleico não emparelhado, análogo à metade de uma escada dividida no meio. Os genomas de fita dupla consistem em dois ácidos nucleicos emparelhados complementares, análogos a uma escada. As partículas virais de algumas famílias de vírus, como as pertencentes aos Hepadnaviridae, contêm um genoma que é parcialmente de fita dupla e parcialmente de fita simples.[56]
Mutação genética
[editar | editar código-fonte]
Os vírus sofrem alterações genéticas por vários mecanismos. Isso inclui um processo chamado desvio antigênico, em que bases individuais no DNA ou RNA se transformam em outras bases. A maioria dessas mutações pontuais é "silenciosa" - elas não alteram a proteína que o gene codifica - mas outras podem conferir vantagens evolutivas, como resistência a medicamentos antivirais.[57][58]
A mudança antigênica ocorre quando há uma grande mudança no genoma do vírus. Isso pode ser resultado de recombinação ou rearranjo. Quando isso acontece com os vírus influenza, podem ocorrer pandemias.[59] Os vírus de RNA geralmente existem como quasispecies ou enxames de vírus da mesma espécie, mas com sequências de nucleosídeos genoma ligeiramente diferentes. Tais quasispecies são um alvo principal para a seleção natural.[60]
Estrutura
[editar | editar código-fonte]Dentre os vários grupos de vírus existentes, não existe um padrão único de estrutura viral. A estrutura mais simples apresentada por um vírus consiste de uma molécula de ácido nucleico coberta por muitas moléculas de proteínas idênticas. Os vírus mais complexos podem conter várias moléculas de ácido nucleico assim como diversas proteínas associadas, envoltório proteico com formato definido, além de complexo envelope externo com espículas. A maioria dos vírus apresentam conformação helicoidal ou isométrica. Dentre os vírus isométricos, o formato mais comum é o de simetria icosaédrica.[1]
Partícula
[editar | editar código-fonte]Os vírus são formados por um agregado de moléculas mantidas unidas por forças secundárias, formando uma estrutura denominada partícula viral. Uma partícula viral completa é denominada vírion. Este é constituído por diversos componentes estruturais (ver tabela abaixo para mais detalhes):[1][2]
- Ácido nucleico: molécula de DNA ou RNA que constitui o genoma viral;
- Capsídeo: envoltório proteico que envolve o material genético dos vírus;
- Nucleocapsídeo: estrutura formada pelo capsídeo associado ao ácido nucleico que ele engloba (Os capsídeos formados pelos ácidos nucleicos são englobados a partir de enzimas);
- Capsômeros: subunidades proteicas (monômeros) que agregadas constituem o capsídeo;
- Envelope: membrana rica em lipídios que envolve a partícula viral externamente. Deriva de estruturas celulares, como membrana plasmática e organelas;
- Peplômeros (espículas): estruturas proeminentes, geralmente constituídas de glicoproteínas e lipídios, que são encontradas ancoradas ao envelope, expostas na superfície.
Morfologia
[editar | editar código-fonte]Abaixo estão listadas as estruturas de vírions mais comuns:
- Legenda dos esquemas: █ Molécula de DNA — █ Molécula de RNA — █ Capsômeros do capsídeo — █ Envelope viral — █ Peplômeros (espículas) — █ Fibras
 |
Vírus icosaédricos não envelopados estão entre os mais comuns. Eles possuem genomas constituídos por dsDNA, ssDNA, dsRNA ou (+)ssRNA. São capazes de infectar organismos de todos os grupos de seres vivos, com exceção de Archaea. Possuem diâmetro que varia de 18 a 60 ηm, compreendendo os menores vírus conhecidos.[4][61] | 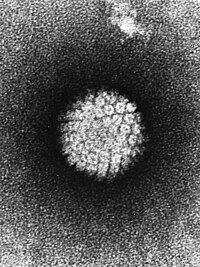 |
 |
Vírus icosaédricos envelopados possuem material genético formado por dsDNA, dsRNA, ou (+)ssRNA. As partículas virais destes vírus possuem diâmetro que varia de 42 a 200 ηm.[4] Vírions icosaédricos envelopados são pouco comuns entre os vírus de animais, sendo observados principalmente nas famílias Arteriviridae, Flaviviridae, Herpesviridae ou Togaviridae. Nenhum vírus de plantas conhecido possui esta estrutura de partícula viral.[61] |  (família: Herpesviridae) |
 |
Partículas virais helicoidais não envelopadas são mais comuns entre vírus que infectam plantas, os quais possuem genoma de ssRNA.[62] Esta é a morfologia do vírus do mosaico do tabaco (TMV), um dos objetos de estudo mais clássicos da virologia, sendo o primeiro vírus a ser descoberto.[63] Além dos vírus de plantas, as famílias Inoviridae (ssDNA) e Rudiviridae (dsDNA), que infectam bactérias e archaea, respectivamente, também possuem esta morfologia. Vírus helicoidais não envelopados tem estrutura em forma de bastão rígido (ver esquema à esquerda), ou de filamento sinuoso (semelhantes ao nucleocapsídeo do esquema abaixo). O comprimento dos vírions varia de 46 ηm (para bastões) a 2 200 ηm (em partículas filamentosas).[4][61] |  |
 |
A morfologia helicoidal envelopada é encontrada principalmente entre vírus (-)ssRNA, entre os quais se encontram muitos agentes etiológicos de doenças humanas conhecidas, como: sarampo (Paramyxoviridae), gripe (Orthomyxoviridae), raiva (Rhabdoviridae), ebola (Filoviridae), hantavirose (Bunyaviridae), febre de Lassa (Arenaviridae). Porém, existem exemplos de vírus com esta conformação que contém material genético composto por dsDNA e (+)ssRNA. Vírus helicoidais envelopados possuem comprimento variando de 60 a 1 950 ηm. Estes vírus podem apresentar formato esférico, filamentoso ou de "bala de revólver" (imagem à direita).[4][61] |  |
 |
O exemplo mais conhecido de vírus de morfologia complexa são os bacteriófagos (ou simplesmente, fagos). O fagos possuem partícula viral composta por uma "cabeça" (capsídeo), de simetria icosaédrica, e uma cauda helicoidal. A cabeça é isométrica ou alongada (50 - 110 ηm de diâmetro), e a cauda pode ser longa e contrátil (Myoviridae: 80 - 455 ηm), longa não contrátil (Siphoviridae: 65 - 570 ηm), ou curta não contrátil (Podoviridae: 17 ηm). Na extremidade da cauda frequentemente são encontradas fibras proteicas que medeiam o contato vírus-célula.[64] Fagos infectam exclusivamente bactérias ou archaea[4] e todos possuem genoma constituído por dsDNA.[5]
Além dos fagos, existem outras famílias virais que possuem vírions com características que contrastam com as morfologias mais usuais, tais como as ilustradas anteriormente. Entre estas famílias estão: Baculoviridae, Reoviridae e Poxviridae.[4][61] |
 |
Ciclo de replicação
[editar | editar código-fonte]Como já mencionado anteriormente, vírus são parasitas intracelulares obrigatórios, pois necessitam do ambiente intracelular de um organismo vivo para se reproduzir. Ao processo de reprodução de um vírus dá-se o nome de replicação viral. O tempo de duração do ciclo de replicação viral varia entre as diversas famílias de vírus, podendo levar poucas horas ou até dias.[65] Esta seção apresentará as etapas envolvidas num ciclo de replicação viral, focado principalmente em vírus que infectam animais. De uma maneira geral, a replicação pode ser dividida em 7 etapas:[4]

Adsorção do vírus à célula
[editar | editar código-fonte]Uma etapa essencial à reprodução viral é a adsorção (ligação) do vírion a uma célula suscetível. A adsorção viral se dá por meio da interação entre proteínas virais, presentes no envelope ou no capsídeo, e receptores celulares que se encontram ancorados a membrana plasmática, expostos ao ambiente extracelular.[66] A ligação entre alguns vírus e células também pode envolver a participação de correceptores (receptores secundários). A especificidade destas interações é alta, como em um modelo chave-fechadura, e determina o tropismo viral para infectar determinadas células e tecidos específicos. Ligações químicas não covalentes, tais como pontes de hidrogênio, atrações iônicas e forças de van der Waals, são responsáveis pela adesão entre as proteínas virais e os receptores celulares.[67]
Nos momentos iniciais da adsorção, a partícula viral interage com um ou poucos receptores, caracterizando uma ligação reversível. Porém, à medida que mais receptores se associam ao vírion, esta ligação passa a ser irreversível, possibilitando a posterior entrada do vírus na célula.[67] Os receptores em geral são proteínas ou carboidratos presentes em glicoproteínas e glicolipídios. Muitas das proteínas receptoras são imunoglobulinas, transportadores transmembrana e canais, ou seja, são estruturas produzidas pelas células para executar funções comuns e essenciais ao bom funcionamento celular. Muitas funcionam como receptores de quimiocinas e fatores de crescimento, ou são responsáveis pelo contato e adesão célula a célula. Os vírus subvertem o papel primordial destas moléculas, utilizando-as como meio para adentrar nas células hospedeiras.[4][5]
Entrada no citosol
[editar | editar código-fonte]Uma vez aderidos à membrana celular, os vírus devem introduzir seu material genético no interior da célula, a fim de que este seja processado (transcrito, traduzido, replicado). Este processo envolve a entrada (penetração) do vírion no citosol e posterior desmontagem do capsídeo para liberação (desnudamento) do genoma viral.[65] Para alcançar o ambiente intracelular, cada vírus utiliza um mecanismo particular. Entre os principais mecanismos (veja imagem abaixo), estão:
- Endocitose: Após a adsorção, a partícula viral pode penetrar no citoplasma por meio de um processo denominado endocitose mediada por receptores, pela formação de endossomos (vesículas). Quando um vírus entra por endocitose, o seu vírion encontra-se envolto pela membrana vesicular. Vírus envelopados liberam os nucleocapsídeos de dentro dos endossomo promovendo a fusão entre o envelope viral e a membrana da vesícula.[5] Já os vírus não envelopados, por não possuírem envelope, utilizam outras estratégias para sair dos endossomos: alguns, como os adenovírus, provocam a lise do endossomo, enquanto outros, como os poliovírus, geram poros na membrana vesicular e injetam o genoma viral diretamente no citosol;[62]
- Fusão: Neste mecanismo, executado apenas por vírus envelopados, o nucleocapsídeo é liberado no interior da célula mediante a fusão entre o envelope viral e a membrana celular. A entrada por fusão pode ocorrer de duas formas: (1) direta, pela fusão do envelope viral com a membrana plasmática, a partir do meio extracelular, ou (2) indireta, sofrendo uma endocitose inicial com posterior fusão já no interior da célula, como citado anteriormente;
- Translocação: por meio da ação de uma proteína receptora, o vírion pode atravessar a membrana por meio de translocação, do ambiente extracelular para o citosol. Este mecanismo é raro e pouco entendido.[4]
Desnudamento do ácido nucleico
[editar | editar código-fonte]Após o processo de penetração, assim que os nucleocapsídeos alcançam o citosol, estes são transportados pelo citoesqueleto (dentro de vesículas ou na forma de nucleocapsídeos livres) em direção ao local específico de processamento do genoma viral, que pode ser no próprio citosol ou no núcleo celular. Para que o genoma possa ser transcrito, traduzido, e replicado, o material genético do vírus deve ser previamente liberado e exposto no ambiente intracelular.[4] A este processo dá-se o nome de desnudamento (ou decapsidação), um procedimento no qual o capsídeo é desmontado completamente ou parcialmente (veja imagem acima). O desnudamento pode ocorrer simultaneamente à entrada do vírus, ou pode acontecer em instantes posteriores. O sítio celular de desnudamento é bastante variável entre as diversas famílias de vírus, podendo ocorrer no citosol (e.g. Togavírus), no interior do endossomo (e.g. Picornavírus), nos poros nucleares (e.g. Adenovírus, Herpesvírus), no interior do núcleo (e.g. Parvovírus, Polyomavírus), ou simplesmente pode não ocorrer (e.g. Reovírus, Poxvírus).[41]
Transcrição e tradução da informação genética
[editar | editar código-fonte]Síntese de mRNA
[editar | editar código-fonte]Como mencionado anteriormente, o Sistema de Classificação de Baltimore foi criado com base nos diferentes mecanismos de transcrição que os vírus adotam para sintetizar mRNA a partir dos seus variados tipos de material genético. Os vírus podem ter genoma constituído por dsDNA, ssDNA, dsRNA, ssRNA, além de alguns serem capazes de realizar a transcrição reversa (ssRNA-RT e dsDNA-RT).[4] Outra propriedade notável dos ácidos nucleicos virais é a polaridade (sentido, ou senso) das fitas de DNA e RNA. Fitas senso positivo (+) apresentam sequência idêntica à do mRNA, enquanto as senso negativo (-) apresentam sequência nucleotídica complementar. Diante desta complexidade de características, as estratégias de transcrição do genoma viral são tão variadas quanto os mecanismos de entrada, e podem envolver mais de uma etapa, as quais levam à conversão da informação genética viral em mRNA.[5]
- Grupo I (dsDNA): Vírus de DNA dupla fita apresentam ORFs em ambas as fitas de DNA, as quais servem diretamente como moldes para a síntese de mRNA. Vírus do grupo I que transcrevem o DNA no interior do núcleo utilizam RNA polimerase II celular para a síntese de mRNA, já aqueles que executam este processo no citosol devem possuir sua própria RNA polimerase DNA-dependente (RpDd) para produzir os transcritos.[4][62]

- Grupo II (ssDNA): Vírus de DNA fita simples apresentam fita positiva ou negativa. Para a síntese de mRNA, estes vírus produzem uma respectiva fita complementar ao seu genoma, gerando uma dupla fita que serve como molde para a transcrição. Estes procedimentos ocorrem no núcleo, com o auxílio de enzimas celulares (RpDd e DpDd (DNA polimerase DNA-dependente)).[4]

- Grupo III (dsRNA): Vírus de RNA dupla fita apresentam uma fita positiva e outra negativa. A fita negativa é utilizada como molde para a síntese de mRNA, em processo que ocorre no citosol, com auxílio de uma RNA polimerase RNA-dependente (RpRd).[4][68]

- Grupo IV ((+)ssRNA): Vírus de RNA fita simples senso positivo apresentam genoma com sequência idêntica à do mRNA, e podem ser utilizados prontamente para a síntese de proteínas. No entanto, é usual a síntese de novas cópias positivas do genoma, mediante a ação de uma RpRd, que produz uma fita negativa que serve como molde para a síntese de novas fitas positivas (mRNAs).[4][5]

- Grupo V ((-)ssRNA): Vírus de RNA fita simples senso negativo, por possuírem genoma com sequência complementar ao mRNA, servem diretamente como molde para a produção de fitas senso positivo. A maioria dos vírus (-)ssRNA (e.g. Rhabdovírus, Filovírus, Bunyavírus, Arenavírus) normalmente procede a transcrição no citosol. Algumas exceções, como os Orthomixovírus, transcrevem seu material genético no núcleo.[4][68]

- Grupo VI (ssRNA-RT): Vírus de RNA com transcrição reversa apresentam genoma de senso positivo. Por meio de uma enzima denominada transcriptase reversa (uma DNA polimerase RNA-dependente), os retrovírus produzem uma fita simples de DNA senso negativo que posteriormente serve de molde à síntese de uma fita positiva de DNA. Ao final, este processo gera uma fita dupla de DNA, que poderá ser integrada ao genoma do hospedeiro no núcleo, e utilizada para a síntese de mRNA viral.[5][62]

- Grupo VII (dsDNA-RT): Vírus de DNA com transcrição reversa (e.g. Hepadnavírus) são vírus dsDNA que promovem a síntese de mRNA no núcleo, sob a ação da RNA polimerase II celular. Neste grupo, a transcrição reversa não ocorre antes síntese de mRNA, como observado nos retrovírus, mas sim posteriormente a replicação do genoma viral.[41][62]

Síntese de proteínas
[editar | editar código-fonte]
1. Transporte do genoma (DNA ou RNA) para o sítio de processamento (núcleo ou citosol)
2. Transcrição (síntese de mRNA)
3. Síntese de proteínas não estruturais
4. Replicação do material genético
5. Síntese de proteínas estruturais
6. Montagem dos nucleocapsídeos
7. Vesícula com glicoproteínas direcionadas ao complexo de Golgi
8. Transporte das proteínas de envelope à membrana plasmática
9. Liberação de partículas virais por lise (vírus não envelopados), ou por brotamento (vírus envelopados)
As proteínas virais são sintetizadas pela maquinaria celular (ribossomos e tRNAs). O processo de tradução ocorre no citosol, em ribossomos livres ou associados ao retículo endoplasmático. Algumas das proteínas sintetizadas em ribossomos livres são transportadas para o núcleo. Proteínas produzidas em ribossomos associados ao retículo são transportadas desta organela para o complexo de Golgi, onde podem sofrer modificações pós-traducionais (glicosilação e fosforilação). O destino final de muitas destas proteínas é a membrana celular, onde estas se concentram em regiões específicas. Em estágios finais da infecção, estas farão parte do envelope de partículas virais que sairão por brotamento nessas regiões.[5][41] Dentro do ciclo de replicação, os primeiros produtos gênicos sintetizados são proteínas não estruturais, como proteínas de ligação ao DNA e enzimas. Entre estas enzimas estão as polimerases e outras moléculas catalíticas, as quais são componentes essenciais à replicação do genoma viral.[69] Já as proteínas estruturais, que formarão as novas partículas virais, normalmente são sintetizadas tardiamente no ciclo de infecção. As novas cópias de material genético sintetizadas são utilizadas para a síntese de mRNAs, os quais codificarão proteínas estruturais que a partir de então serão produzidas em grandes quantidades para compor os vírus em formação.[70] Os diferentes vírus de DNA e RNA possuem mecanismos próprios de regulação da expressão gênica, os quais controlam a produção de proteínas em momentos e quantidades apropriadas às necessidades virais.[71]
Replicação do genoma viral
[editar | editar código-fonte]Na maioria dos casos, o genoma é replicado no mesmo local onde ocorre a transcrição do material genético do vírus, isto é, no citoplasma ou no núcleo.[4] Assim como ocorre na transcrição, o processo de replicação de genomas virais envolve a participação de polimerases. Vírus de fita simples, dos grupos II, IV e V, precisam produzir uma fita complementar ao genoma, que posteriormente servirá de molde para a síntese do material genético.[72] Vírus de fita dupla, dos grupos I e III, utilizam cada uma das duas fitas para gerar suas respectivas cópias complementares. Em geral, moléculas de DNA são sintetizadas a partir de outras moléculas de DNA (DNA → DNA), e o mesmo acontece com moléculas de RNA (RNA → RNA). A exceção a esta regra fica por conta dos vírus que realizam transcrição reversa.[73] Membros do grupo VI (ssRNA-RT) replicam o seu genoma a partir de um intermediário de DNA (RNA → DNA → RNA). Já os membros do I (dsDNA-RT) replicam o seu genoma a partir de um intermediário de RNA (DNA → RNA → DNA).[5]
Montagem do vírion
[editar | editar código-fonte]A montagem corresponde ao processo de formação das partículas virais infectivas (vírions). Neste estágio do ciclo de infecção, as proteínas estruturais sintetizadas em etapas anteriores se associam para constituir o capsídeo. Capsídeos com formato helicoidal são formados em torno da superfície da molécula de ácido nucléico.[69] Já os capsídeos de simetria icosaédrica são montados previamente e depois preenchidos com o genoma viral, através de um poro na estrutura pré-formada denominada pró-capsídeo. O pró-capsídeo de alguns vírus pode sofrer modificações que levam a formação do capsídeo maduro.[5] O sítio de montagem dos capsídeos depende do local de replicação viral na célula, e varia entre as diversas famílias de vírus.[4] O procedimento de montagem de vírus não envelopados se resume a formação dos nucleocapsídeos, enquanto que para vírus envelopados a montagem só se finaliza depois da aquisição do envelope viral. A membrana lipídica do envelope se origina a partir de estruturas celulares, como: membrana plasmática (e.g. Paramyxovírus, Orthomixovírus, Rhabdovírus) e compartimentos membranosos intracelulares (complexo de Golgi, retículo endoplasmático, núcleo).[65] Outro mecanismo de aquisição de envelope é a denominada “síntese de novo” de membranas, no qual o envelope é gradualmente construído em volta do nucleocapsídeo. Este processo pode ocorrer no núcleo (e.g. Baculovírus) ou no citoplasma (e.g. Poxvírus).[5]
Liberação de novas partículas virais
[editar | editar código-fonte]A liberação dos vírions do citosol pode se dar por lise celular ou brotamento. A liberação por lise celular é mais comum aos vírus não envelopados, e ocorre quando a membrana plasmática da célula infectada se rompe, levando-a morte celular.[4] Porém, nem todo processo de liberação viral causa danos a célula hospedeira. O brotamento é um mecanismo de liberação que pode provocar pouco ou nenhum prejuízo à célula.[41] Vírus que obtém envelope a partir da membrana plasmática saem da célula por meio de brotamento direto do nucleocapsídeo em contato com a face interna da membrana, em regiões específicas, onde se localizam as glicoproteínas virais sintetizadas em momentos prévios da infecção. Vírus com envelope originado de compartimentos intracelulares (organelas) são liberados da célula por meio de vesículas que se fundem com a membrana plasmática. Após a liberação, quando os vírions se encontram no meio extracelular, a maioria deles permanece inerte até que outra célula hospedeira seja infectada, reiniciando o ciclo de replicação viral.[5]
Doenças humanas virais
[editar | editar código-fonte]Assim como muitos parasitas, os vírus são patogênicos aos seres vivos. Ao invadirem as células de um indivíduo, eles prejudicam o funcionamento normal dessas células e, consequentemente, provocam doenças.[74] Entre as principais viroses humanas estão: gripe, hepatite (A, B e C), caxumba, sarampo, varicela (catapora), SIDA (AIDS), raiva, dengue, febre amarela, poliomielite (paralisia infantil), rubéola, meningite, encefalite, herpes, pneumonia, entre outras doenças.[75] Recentemente foi mostrado que o câncer cervical é causado ao menos em partes pelo papilomavírus (que causa papilomas, ou verrugas), representando a primeira evidência significante em humanos para uma ligação entre câncer e agentes virais.[76]
Epidemiologia
[editar | editar código-fonte]A epidemiologia viral é o ramo da ciência médica que lida com a transmissão e o controle de infecções por vírus em humanos. A transmissão de vírus pode ser vertical, o que significa de mãe para filho, ou horizontal, o que significa de pessoa para pessoa.[77] Exemplos de transmissão vertical incluem o vírus da hepatite B e o HIV, onde o bebê já nasceu infectado pelo vírus. Outro exemplo, mais raro, é o vírus da varicela zoster, que, apesar de causar infecções relativamente leves em crianças e adultos, pode ser fatal para o feto e o bebê recém-nascido.[78]
A transmissão horizontal é o mecanismo mais comum de disseminação de vírus nas populações. A transmissão pode ocorrer quando: os fluidos corporais são trocados durante a atividade sexual, por exemplo, HIV; o sangue é trocado por transfusão contaminada ou compartilhamento de agulhas, por exemplo, hepatite C; troca de saliva pela boca, por exemplo, vírus Epstein-Barr; comida ou água contaminada é ingerida, por exemplo, norovírus; aerossóis contendo virions são inalados, por exemplo, vírus influenza; e vetores de insetos, como mosquitos, penetram na pele de um hospedeiro, por exemplo, dengue.[79] A taxa ou velocidade de transmissão de infecções virais depende de fatores que incluem a densidade populacional, o número de indivíduos suscetíveis (ou seja, aqueles que não são imunes), a qualidade dos cuidados com a saúde e o clima.[80]
A epidemiologia é usada para quebrar a cadeia de infecção nas populações durante surtos de doenças virais. São usadas medidas de controle baseadas no conhecimento de como o vírus é transmitido. É importante encontrar a fonte ou fontes do surto e identificar o vírus.[81] Uma vez identificado o vírus, às vezes a cadeia de transmissão pode ser interrompida pelas vacinas. Quando as vacinas não estão disponíveis, o saneamento e a desinfecção podem ser eficazes. Frequentemente, as pessoas infectadas são isoladas do resto da comunidade e as que foram expostas ao vírus são colocadas em quarentena.[51]
Epidemias e pandemias
[editar | editar código-fonte]
Uma pandemia é uma epidemia mundial. A Gripe espanhola, que durou até 1919, foi uma pandemia de gripe da categoria 5 causada por um vírus da gripe A incomumente grave e mortal. As vítimas eram frequentemente jovens adultos saudáveis, em contraste com a maioria dos surtos de gripe, que afetam predominantemente pacientes jovens, idosos ou enfraquecidos.[82] Estimativas mais antigas dizem que matou de 40 a 50 milhões de pessoas,[83] enquanto pesquisas mais recentes sugerem que pode ter matado até 100 milhões de pessoas, ou 5% da população mundial em 1918.[84][85]
Embora as pandemias virais sejam eventos raros, o HIV - que evoluiu a partir de vírus encontrados em macacos e chimpanzés - tem sido uma pandemia desde pelo menos a década de 1980.[86] Durante o século XX, houve quatro pandemias causadas pelo vírus influenza e as que ocorreram em 1918, 1957 e 1968 foram graves.[87] A maioria dos pesquisadores acredita que o HIV se originou na África Subsariana durante o século XX;[88] agora é uma pandemia, com cerca de 37,9 milhões de pessoas vivendo com a doença no mundo todo. Houve cerca de 770 mil mortes por AIDS em 2018. O Programa Conjunto das Nações Unidas sobre HIV/AIDS (UNAIDS) e a Organização Mundial da Saúde (OMS) estimam que a AIDS matou mais de 25 milhões de pessoas desde que foi reconhecida pela primeira vez em 5 Junho de 1981, tornando-a uma das epidemias mais destrutivas da história registrada.[89] Em 2007, houve 2,7 milhões de novas infecções por HIV e 2 milhões de mortes relacionadas ao HIV.[90]

Vários patógenos virais altamente letais são membros dos Filoviridae. Filovírus são vírus do tipo filamento que causam febre hemorrágica viral e incluem ebolavírus e marburgvírus. O vírus Marburg, descoberto pela primeira vez em 1967, atraiu a atenção da imprensa em abril de 2005 para um surto em Angola.[91] A doença pelo vírus Ebola também causou surtos intermitentes com altas taxas de mortalidade desde 1976, quando foi identificado pela primeira vez. A pior e mais recente é a epidemia de 2013-2016 na África Ocidental.[92]
A síndrome respiratória aguda grave (SARS) e a síndrome respiratória do Oriente Médio (MERS) são causadas por novos tipos de coronavírus. Sabe-se que outros coronavírus causam infecções leves em seres humanos,[93] então a virulência e a rápida disseminação de infecções por SARS - que em julho de 2003 haviam causado cerca de 8 mil casos e 800 mortes - eram inesperadas e a maioria dos países não estava preparada.[94]

Um coronavírus relacionado surgiu em Wuhan, China em novembro de 2019 e se espalhou rapidamente pelo mundo. Pensa-se que tenha se originado em morcegos e posteriormente chamado de síndrome respiratória aguda grave como coronavírus 2, infecções pelo vírus causaram uma pandemia em 2020.[95][96] Restrições sem precedentes em tempos de paz foram impostas a viagens internacionais e o toque de recolher imposto em várias grandes cidades do mundo.[97]
Câncer
[editar | editar código-fonte]
Os vírus são uma causa estabelecida de câncer em humanos e outras espécies. O câncer viral ocorre apenas em uma minoria de pessoas infectadas (ou animais). Os vírus do câncer provêm de uma variedade de famílias de vírus, incluindo vírus de RNA e DNA, e, portanto, não existe um tipo único de "oncovírus" (um termo obsoleto originalmente usado para retrovírus de transformação aguda). O desenvolvimento do câncer é determinado por uma variedade de fatores, como imunidade ao hospedeiro[98] e mutações no hospedeiro.[99]
Os vírus aceitos para causar câncer em humanos incluem alguns genótipos de papilomavírus humano, vírus da hepatite B, vírus da hepatite C, vírus Epstein-Barr, herpesvírus associado ao sarcoma de Kaposi e vírus linfotrópico T humano.[100] O vírus do câncer humano descoberto mais recentemente é um poliomavírus (poliomavírus de células Merkel) que causa a maioria dos casos de uma forma rara de câncer de pele chamada carcinoma de células Merkel.[101]
Prevenção e tratamento de doenças virais
[editar | editar código-fonte]Devido ao uso das células do hospedeiro, os vírus tornam-se difíceis de se combater. Como os tratamentos quimioterápicos para a infecções virais são limitados, os tratamentos sintomáticos, como descanso, hidratação e analgésicos, são as alternativas mais comuns para reduzir os incômodos causados pela maioria das doenças virais, principalmente infecções respiratórias. Pesquisas realizadas com camundongos infectados com o vírus coxsackie B demonstraram que esforços físicos severos, repetitivos e exaustivos prolongaram a infecção e provocaram o retardo do início da resposta imune via interferons e anticorpos.[75]
Quando as células são atacadas por vírus, o sistema de defesa do organismo parasitado passa a produzir anticorpos específicos que combatem o vírus invasor. Isso ocorre porque os vírus são formados por proteínas diferentes das do organismo parasitado. Estas proteínas são reconhecidas como não próprias do organismo e são neutralizadas pelos anticorpos. Assim, caso o mesmo vírus invada o organismo novamente, a memória imunológica desencadeará rapidamente uma resposta imune específica contra o vírus, e a doença não se instalará.[67]
Vacina virais
[editar | editar código-fonte]
As vacinas são soluções médicas eficazes para prevenir algumas infecções virais. Elas podem ser produzidas a partir de vírus inativados ou atenuados, ou a partir de subunidades de proteínas virais. Uma vez introduzidos num indivíduo, os componentes das vacinas são capazes de estimular o organismo a produzir uma resposta imunológica humoral e/ou celular.[102] O indivíduo desenvolve memória imunológica quando é exposto uma ou algumas vezes aos antígenos presentes na vacina. A vacinação é empregada com o objetivo de prevenir a manifestação de doenças virais futuras. Portanto, vacinas não são aplicadas com o intuito de curar viroses já instaladas, mas sim para evitar o desenvolvimento da doença.[74]
A vacina Sabin, usada para prevenir a poliomielite (ou paralisia infantil), é uma das vacinas virais atenuadas mais amplamente utilizadas no mundo. Testes com macacos demonstraram que o vírus atenuado, diferentemente da cepa viral patogênica, não possui virulência contra os tecidos nervosos do cérebro e da medula espinhal. Porém, como o organismo não diferencia um vírus do outro, ele passa a produzir os anticorpos necessários, imunizando o indivíduo vacinado contra o vírus da poliomielite.[103]
Drogas antivirais
[editar | editar código-fonte]
As drogas antivirais são substâncias utilizadas no tratamento específico contra determinados vírus. Entre as principais substâncias antivirais, estão: o aciclovir (contra o herpesvírus), a ribavirina (contra o vírus da hepatite C), o oseltamivir (contra o vírus da gripe), o ritonavir, o indinavir, a zidovudina, entre outras, contra o vírus da AIDS (SIDA).[104] Diferentemente do que ocorre nos casos de infecções bacterianas, os antibióticos não são úteis contra infecções virais. O uso abusivo e inadequado de antibióticos, como contra infecções virais, tem se tornado um grave problema de saúde pública por ser uma das causas do recorrente surgimento de bactérias resistentes a múltiplos antibióticos.[105]
Infecção em outras espécies
[editar | editar código-fonte]Os vírus infectam toda a vida celular e, embora os vírus ocorram universalmente, cada espécie celular tem seu próprio intervalo específico que geralmente infecta apenas essas espécies.[65] Alguns vírus, chamados satélites, podem se replicar apenas dentro de células que já foram infectadas por outro vírus.[106]
Vírus de planta
[editar | editar código-fonte]Existem muitos tipos de vírus de plantas, mas geralmente causam apenas uma perda de rendimento, e não é economicamente viável tentar controlá-los. Os vírus das plantas são frequentemente transmitidos de planta para planta por organismos, conhecidos como vetores. Geralmente são insetos, mas alguns fungos, vermes de nematóides e organismos unicelulares têm se mostrado vetores.[107][108]
Quando o controle de infecções por vírus de plantas é considerado econômico, para frutos perenes, por exemplo, os esforços estão concentrados na morte dos vetores e na remoção de hospedeiros alternativos, como as ervas daninhas. Os vírus vegetais não podem infectar seres humanos e outros animais porque eles podem se reproduzir apenas nas células vegetais vivas.[109][110]
Vírus de bactéria
[editar | editar código-fonte]
Os bacteriófagos são um grupo comum e diversificado de vírus e são a entidade biológica mais abundante em ambientes aquáticos - há até dez vezes mais desses vírus nos oceanos do que as bactérias,[111] atingindo níveis de 250 000 000 bacteriófagos por mililitro de água do mar. Esses vírus infectam bactérias específicas ligando-se às moléculas receptoras da superfície e entrando na célula.[112]
Dentro de um curto período de tempo, em alguns casos, apenas alguns minutos, a polimerase bacteriana começa a traduzir o mRNA viral em proteína. Essas proteínas tornam-se novos virions dentro da célula,[111] proteínas auxiliares, que ajudam na montagem de novos virions, ou proteínas envolvidas na lise celular. As enzimas virais auxiliam na quebra da membrana celular e, no caso do fago T4, em pouco mais de vinte minutos após a injeção, mais de trezentos fagos podem ser liberados.[113]
Vírus de animais
[editar | editar código-fonte]Os vírus são patógenos importantes do gado. Doenças como febre aftosa e febre catarral ovina são causadas por vírus.[114] Animais de companhia, como gatos, cães e cavalos, se não forem vacinados, são suscetíveis a infecções virais graves. O parvovírus canino é causado por um pequeno vírus de DNA e as infecções geralmente são fatais em filhotes.[115] Como todos os invertebrados, a abelha é suscetível a muitas infecções virais. A maioria dos vírus coexiste inofensivamente no hospedeiro e não causa sinais ou sintomas de doença.[116]
Vírus de arqueias
[editar | editar código-fonte]Alguns vírus se replicam dentro das arqueias: são vírus de DNA de fita dupla com formas incomuns e às vezes únicas.[117] Esses vírus foram estudados com mais detalhes nas arqueias termofílicas, particularmente as ordens Sulfolobales e Termoproteales.[118]
As defesas contra esses vírus envolvem interferência de RNA de sequências repetitivas de DNA em genomas arcaicos que estão relacionados aos genes dos vírus.[119][120] A maioria das arqueias possui sistemas CRISPR-Cas como defesa adaptativa contra vírus. Isso permite que a arqueia retenha seções do DNA viral, que são usadas para direcionar e eliminar infecções subsequentes pelo vírus, usando um processo semelhante à interferência do RNA.[121]
Vírus no ecossistema aquático
[editar | editar código-fonte]
Os vírus são a entidade biológica mais abundante em ambientes aquáticos. Existem cerca de dez milhões deles em uma colher de chá de água do mar.[122] A maioria desses vírus são bacteriófagos que infectam bactérias heterotróficas e cianófagos que infectam cianobactérias e são essenciais para a regulação dos ecossistemas de água salgada e de água doce. Os bacteriófagos são inofensivos para plantas e animais e são essenciais para a regulação dos ecossistemas marinhos e de água doce são importantes agentes de mortalidade do fitoplâncton, a base da cadeia alimentar em ambientes aquáticos.[123][124]
Eles infectam e destroem bactérias em comunidades microbianas aquáticas e são um dos mecanismos mais importantes de reciclagem do ciclo de carbono e nutrientes em ambientes marinhos. As moléculas orgânicas liberadas pelas células bacterianas mortas estimulam o crescimento bacteriano e algal fresco, em um processo conhecido como desvio viral.[125] Em particular, a lise de bactérias por vírus demonstrou melhorar o ciclo do nitrogênio e estimular o crescimento do fitoplâncton. A atividade viral também pode afetar a bomba biológica, o processo pelo qual o carbono é sequestrado no oceano profundo.[126]
Os microrganismos constituem mais de 90% da biomassa no mar. Estima-se que os vírus matem aproximadamente 20% dessa biomassa por dia e que haja 10 a 15 vezes mais vírus nos oceanos do que bactérias e archaea.[127] Os vírus também são os principais agentes responsáveis pela destruição do fitoplâncton, incluindo a proliferação de algas prejudiciais, o número de vírus nos oceanos diminui ainda mais no mar e mais fundo na água, onde há menos organismos hospedeiros.[128][129]
Origem da diversidade genética viral
[editar | editar código-fonte]Diversos são os processos responsáveis por gerar variabilidade genética dentro de uma população viral. Entre tais processos, estão: mutações, recombinações, rearranjos genéticos em coinfeções, entre outros. A fidelidade e a frequência dos processos de replicação, as taxas de ocorrência de coinfecções, o modo de transmissão, o tamanho e a estrutura das populações (virais e de hospedeiros) são fatores que influenciam a geração da variabilidade genética viral.[4] Quando os vírus se reproduzem no interior de uma célula, o material genético viral pode sofrer mutações, originando uma grande diversidade genética a partir de um único tipo de vírus. Vírus de RNA, que dependem das enzimas RNA polimerase ou transcriptase reversa para se replicar, apresentam taxas de mutação mais elevadas, se comparados a vírus de DNA.[130] Isto ocorre porque tais enzimas não são capazes de corrigir os erros provocados no decorrer da replicação. Vírus de DNA, que usam a maquinaria enzimática celular, apresentam taxas reduzidas de mutações genéticas, pois utilizam enzimas celulares que possuem a habilidade de reparar os erros gerados durante a síntese de DNA.[62]
Agentes infecciosos subvirais
[editar | editar código-fonte]Agentes subvirais são partículas infecciosas subcelulares bastante simples estruturalmente que não são enquadradas como vírus no sentido estrito do termo.[131] Entres tais agentes, destacam-se os vírus satélite, os virusóides, os viróides, os RNAs satélite, os RNAs interferentes defectivos (DI-RNAs) e os príons:[132]
- Vírus satélites: são moléculas de DNA ou RNA viral que carecem de informações genéticas essenciais para garantir sua independência replicativa. Vírus satélites dependem de outros vírus (vírus helper) para obter os fatores biológicos (proteínas) necessários para a infecção de uma célula;[132][133]
- Virusóides: são moléculas de ssRNA circular que não codificam proteínas. Dependem de vírus helpers para se replicar e formar capsídeos;[132]
- Viróides: são patógenos de plantas constituídos apenas por moléculas de ssRNA circular, altamente estáveis, as quais não são capazes de codificar nenhuma proteína;[132]

- RNAs satélite: considerados subtipos de virusóides, são constituídos por pequenas moléculas de RNA, que variam de 200 a 1700 nucleotídeos, sendo que os maiores são capazes de codificar algumas proteínas;[132]
- RNAs interferentes defectivos (DI-RNAs): são pequenas moléculas de RNA viral provenientes de genomas virais que perderam função essenciais em decorrência de seguidas deleções. Um DI-RNA depende essencialmente do vírus parental (que o originou) para se replicar;[133]
- Príons (ou priões): são agentes infecciosos que não possuem nenhum ácido nucleico, sendo constituídos exclusivamente por um único tipo de proteína estruturalmente modificada que tem a capacidade de converter proteínas semelhantes e normais em proteínas alteradas quanto a conformação tridimensional. Tais proteínas alteradas se agregam e causam danos em células nervosas.[134]
Aplicações
[editar | editar código-fonte]Ciências da vida e medicina
[editar | editar código-fonte]
Os vírus são importantes para o estudo da biologia molecular e celular, pois fornecem sistemas simples que podem ser usados para manipular e investigar as funções das células.[49] O estudo e o uso de vírus forneceram informações valiosas sobre aspectos da biologia celular. Por exemplo, os vírus têm sido úteis no estudo da genética e ajudaram a entender os mecanismos básicos da genética molecular, como replicação de DNA, transcrição, Modificação pós-transcricional, tradução, transporte de proteínas e imunologia.[135]
A genética usa vírus como vetores para inserir genes nas células que estudam. Isso é útil para fazer com que as células produzam matérias estranhas ou para estudar o efeito da inserção de novos genes no genoma. Da mesma forma, a terapia de vírus usa vírus como vetores para tratar várias doenças, porque pode atingir especificamente células e DNA.[136] Usos promissores são mostrados no câncer e na terapia genética. Os cientistas da Europa Oriental têm usado a fagoterapia como uma alternativa aos antibióticos há algum tempo, e o interesse nessa abordagem está aumentando devido ao alto nível de resistência a antibióticos em algumas bactérias patogênicas.[137]
Armas
[editar | editar código-fonte]A capacidade dos vírus de causar epidemias devastadoras nas sociedades humanas levou à preocupação de que os vírus pudessem ser armados para a guerra biológica. Outra preocupação foi levantada pela recreação bem-sucedida do infame vírus da influenza de 1918 em um laboratório.[138]
O vírus da varíola devastou inúmeras sociedades ao longo da história antes de sua erradicação. Existem apenas dois centros no mundo autorizados pela OMS a manter estoques de vírus da varíola: o VECTOR do Centro Estadual de Pesquisa em Virologia e Biotecnologia na Rússia e os Centros de Controle e Prevenção de Doenças nos Estados Unidos.[139] Pode ser usada como arma, pois a vacina contra a varíola às vezes teve efeitos colaterais graves, não sendo mais usada rotineiramente em nenhum país. Assim, grande parte da população humana moderna quase não tem resistência estabelecida à varíola e seria vulnerável ao vírus.[138][139]
Ciência dos materiais e nanotecnologia
[editar | editar código-fonte]As tendências atuais em nanotecnologia prometem fazer um uso muito mais versátil de vírus. Do ponto de vista de um cientista de materiais, os vírus podem ser considerados nanopartículas orgânicas. Sua superfície carrega ferramentas específicas que lhes permitem atravessar as barreiras de suas células hospedeiras.[140][141]
O tamanho e a forma dos vírus, o número e a natureza dos grupos funcionais em sua superfície são definidos com precisão. Como tal, os vírus são comumente usados na ciência dos materiais como andaimes para modificações de superfície ligadas covalente mente. Uma qualidade específica dos vírus é que eles podem ser adaptados pela evolução direcionada. As poderosas técnicas desenvolvidas pelas ciências da vida estão se tornando a base das abordagens de engenharia para os nanomateriais, abrindo uma ampla gama de aplicações muito além da biologia e da medicina.[141]
Devido ao seu tamanho, forma e estruturas químicas bem definidas, os vírus têm sido usados como modelos para organizar os materiais em nanoescala. Exemplos recentes incluem trabalhos no Naval Research Laboratory em Washington, DC, usando partículas do vírus do mosaico do caupi (CPMV) para amplificar sinais em sensores baseados em microarrays de DNA.[142] Nesta aplicação, as partículas do vírus separam os corantes fluorescentes usados para sinalizar para impedir a formação de dímeros não fluorescentes que atuam como inibidores. Outro exemplo é o uso do CPMV como uma placa de ensaio em nanoescala para eletrônica molecular.[143]
Vírus sintéticos
[editar | editar código-fonte]Muitos vírus podem ser sintetizados de novo ("do zero") e o primeiro vírus sintético foi criado em 2002. Embora seja um equívoco, não é o vírus real que é sintetizado, mas seu genoma de DNA (no caso de um vírus de DNA) ou uma cópia de cDNA de seu genoma (no caso de vírus de RNA). Para muitas famílias de vírus, o DNA ou RNA sintético nu (uma vez convertido enzimaticamente a partir do cDNA sintético) é infeccioso quando introduzido na célula.[144][145]
Ou seja, eles contêm todas as informações necessárias para produzir novos vírus. Essa tecnologia está sendo usada agora para investigar novas estratégias de vacinas. A capacidade de sintetizar vírus tem conseqüências de longo alcance, uma vez que os vírus não podem mais ser considerados extintos, desde que as informações de sua sequência genômica sejam conhecidas e células permissivas estejam disponíveis.[145]
Ver também
[editar | editar código-fonte]Referências
- ↑ a b c d e f FRAENKEL-CONRAT, H.; WILLIAMS, R.C. (1955). «Reconstitution of active tobacco mosaic virus from its inactive protein and nucleic acid components». Proc Natl Acad Sci U S A. 41: 690–698. ISSN 0027-8424. PMC 528165
 . PMID 16589730. doi:10.1073/pnas.41.10.690
. PMID 16589730. doi:10.1073/pnas.41.10.690
- ↑ a b c d e MELNICK, J. L.; JAWETZ, E.; ADELBERG, E. (1998). Microbiologia medica. Rio de Janeiro: Guanabara Koogan: [s.n.] p. 524. 653 páginas
- ↑ Mahy, Brian W. J. (2001). A Dictionary of Virology. [S.l.]: Academic Press. p. 405-406. 422 páginas. ISBN 0124653278
- ↑ a b c d e f g h i j k l m n o p q r s t u v CANN, A. J. (2005). Principles of Molecular Virology. 4. [S.l.]: Massachusetts: Elsevier Academic Press. p. 242-279. 352 páginas. ISBN 0120887878
- ↑ a b c d e f g h i j k l m n o p CARTER, J.; SAUNDERS, V. (2007). Virology: Principles and Applications. [S.l.]: Chichester: Wiley. p. 20-32. 358 páginas. ISBN 9780470023860
- ↑ Fox, Alex (25 de abril de 2019). «The oceans contain nearly 200,000 kinds of viruses». Science | AAAS (em inglês). Consultado em 15 de junho de 2020
- ↑ «Existem mais vírus do que estrelas no universo. Por que apenas alguns nos infectam?». National Geographic. 25 de novembro de 2020. Consultado em 8 de julho de 2021
- ↑ «Virus, n.». OED Online. Oxford University Press
- ↑ Harper D (2011). «virus». The Online Etymology Dictionary
- ↑ Harper D (2011). «virion». The Online Etymology Dictionary
- ↑ Horzinek 1997, p. 15-20.
- ↑ MAYER, A. (1886). «Über die Mosaikkrankheit des Tabaks». Die Landwirtschaftliche Versuchs-stationen (em alemão). 32: 451–467
- ↑ Zaitlin 1998, p. 105–110.
- ↑ Iwanowski 1892, p. 67-70.
- ↑ Beijerinck 1898, p. 1-22.
- ↑ a b Collier, Balows & Sussman 1998, p. 3.
- ↑ Levine, Arnold J.; Enquist, Lynn W.; A. Zanotto, Paolo M. de (2007). «História de Virologia – BMM 0586» (PDF). edisciplinas.usp.br. Consultado em 16 de junho de 2020
- ↑ a b d'HERELLE, F. (1917). «Sur un microbe invisible antagonistic des bacilles dysenterique». C. R. Acad. Sci. Paris. 165: 373-375
- ↑ STANLEY, W.M. (1935). «Isolation of a crystalline protein possessing the properties of tobacco mosaic virus». Science. 81: 644-645
- ↑ KAUSCHE, G.A.; ANKUCH, P.F.; RUSKA, H. (1939). «Die Sichtbarmachung von PF lanzlichem Virus in Ubermikroskop». Naturwissenschaften. 27: 292–299
- ↑ «VIROLOGIA - estudo dos vírus» (PDF). professor.pucgoias.edu.br. Consultado em 16 de junho de 2020
- ↑ ELLERMANN, V.; BANG, O. (1908). «Experimentelle leukamie bei huhnern». Zentralbl Bakteriol Parasitenkd Infektionskr Hyg. 46: 595–597. OCLC 603732629
- ↑ ROUS, P. (1911). «A sarcoma of the fowl transmissible by an agent separable from the tumor cells». J Exp Med. 13: 397–399. PMC 2124874
 . PMID 19867421. doi:10.1084/jem.13.4.397
. PMID 19867421. doi:10.1084/jem.13.4.397
- ↑ a b Vieira & Nahas 2000, p. 539-545.
- ↑ TWORT, F.W. (1915). «An investigation on the nature of ultra-microscopic viruses». Lancet. 2: 1241-1243. doi:10.1016/S0140-6736(01)20383-3
- ↑ Quinn 2005, p. 274.
- ↑ Steinhardt E, Israeli C, Lambert R.A. (1913). «Studies on the cultivation of the virus of vaccinia». J. Inf Dis. 13 (2): 294–300. doi:10.1093/infdis/13.2.294
- ↑ MAITLAND, H.B.; MAITLAND, M.C. (1928). «Cultivation of vaccinia virus without tissue culture». Lancet. 512 (5482): 596-597. doi:10.1016/S0140-6736(00)84169-0
- ↑ GOODPASTURE, W.E.; WOODRUFF, A.M. (1931). «The susceptibility of the chorio-allantoic membrane of chick embryos to infection with the fowl-pox virus». Am J Pathol. 7 (3): 209-222. PMC 2062632
 . PMID 19969963
. PMID 19969963
- ↑ FRIERSON, J.G. (2010). «The Yellow Fever Vaccine: A History». Yale Journal of Biology and Medicine. 83 (2): 77–85. PMC 2892770
 . PMID 20589188
. PMID 20589188
- ↑ ENDERS, J.F.; WELLER, T.H.; ROBBINS, F.C. (1949). «Cultivation of the Lansing strain of poliomyelitis virus in cultures of various human embryonic tissues». Science. 1098: 85–87. PMID 17794160. doi:10.1126/science.109.2822.85
- ↑ Rosen 2004, p. 1481-1483.
- ↑ Welle, Deutsche (9 de março de 2019). «1931: Físico alemão cria microscópio eletrônico». DW. Consultado em 15 de junho de 2020
- ↑ Bawden & Pirie 1937, p. 274-320.
- ↑ BERNAL, J.D.; FANKUCHEN, I. (1941). «X-ray and crstallographic studies of plant virus preparations». J Gen Physiol. 25 (1): 111-146. OCLC 679238809. PMC 2142028
 . PMID 19873256. doi:10.1085/jgp.25.1.147
. PMID 19873256. doi:10.1085/jgp.25.1.147
- ↑ CREAGEN, A.N.; MORGAN, G.J. (2008). «After the double helix: Rosalind Franklin's research on Tobacco mosaic virus». Isis. 99 (2): 239–72. PMID 18702397. doi:10.1086/588626
- ↑ Norrby, E. (2008). «Nobel Prizes and the emerging virus concept». Arch. Virol. 153 (6): 1109–1123. PMID 18446425. doi:10.1007/s00705-008-0088-8. Consultado em 15 de junho de 2020
- ↑ Temin HM, Baltimore D (1972). «RNA-directed DNA synthesis and RNA tumor viruses». Adv. Virus Res. 17: 129–86. PMID 4348509. doi:10.1016/S0065-3527(08)60749-6
- ↑ Barré-Sinoussi, F. (1983). «Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS)». Science. 220 (4599): 868–871. PMID 6189183. doi:10.1126/science.6189183. Consultado em 15 de junho de 2020
- ↑ a b c d e ACHESON, Nicholas. H. (2007). Fundamentals of Molecular Virology. [S.l.]: Chichester: Wiley. p. 26. 432 páginas. ISBN 9780471351511. OCLC 255748189
- ↑ Patton, John T. (2008). Segmented double-stranded RNA viruses : structure and molecular biology. Norfolk, UK: Caister Academic Press. p. 116-120. 373 páginas. ISBN 9781904455219. OCLC 191167139
- ↑ Robertson, Michael P.; Igel, Haller; Baertsch, Robert; Haussler, David; Jr, Manuel Ares; Scott, William G. (28 de dezembro de 2004). «The Structure of a Rigorously Conserved RNA Element within the SARS Virus Genome». PLOS Biology (em inglês). 3 (1): e5. ISSN 1545-7885. PMC 539059
 . PMID 15630477. doi:10.1371/journal.pbio.0030005. Consultado em 15 de junho de 2020
. PMID 15630477. doi:10.1371/journal.pbio.0030005. Consultado em 15 de junho de 2020
- ↑ Levinson, Warren,. Review of medical microbiology and immunology Thirteenth edition ed. New York: [s.n.] p. 230. 800 páginas. ISBN 9780071818117. OCLC 871305336
- ↑ Collier, Reed; Normandy, Georges (1934). «Le Charnier». Books Abroad. 8 (1): 79. ISSN 0006-7431. doi:10.2307/40074967. Consultado em 26 de junho de 2020
- ↑ Collier, Jeremy, 1650-1726. (1708–1714). An ecclesiastical history of Great Britain, chiefly of England : from the first planting of Christianity, to the end of the reign of Charles the Second : with a brief account of the affairs of religion in Ireland : collected from the best ancient historians, councils, and records : in two volumes. 1. [S.l.]: Printed for Samuel Keble ... and Benjamin Tooke. p. 339-342. OCLC 15388245
- ↑ Iyer, Lakshminarayan M.; Balaji, S.; Koonin, Eugene V.; Aravind, L. (1 de abril de 2006). «Evolutionary genomics of nucleo-cytoplasmic large DNA viruses». Virus Research. Comparative Genomics and Evolution of Complex Viruses (em inglês). 117 (1): 156–184. ISSN 0168-1702. PMID 16494962. doi:10.1016/j.virusres.2006.01.009. Consultado em 25 de junho de 2020
- ↑ Sanjuán, Rafael; Nebot, Miguel R.; Chirico, Nicola; Mansky, Louis M.; Belshaw, Robert (1 de outubro de 2010). «Viral Mutation Rates». Journal of Virology (em inglês). 84 (19): 9733–9748. ISSN 0022-538X. PMC 2937809
 . PMID 20660197. doi:10.1128/JVI.00694-10. Consultado em 25 de junho de 2020
. PMID 20660197. doi:10.1128/JVI.00694-10. Consultado em 25 de junho de 2020
- ↑ a b c Powell, Christopher (2019). Fungi, Bacteria and Viruses. [S.l.: s.n.] p. 151-173. 344 páginas. ISBN 9781839471896
- ↑ a b c d Mahy, B. W. J. (Brian W. J.); Van Regenmortel, M. H. V. (2010). Desk encyclopedia of general virology. Oxford, UK: Academic Press. p. 24. 672 páginas. ISBN 9780123751461. OCLC 480587729
- ↑ a b Turner, Raphael (16 de novembro de 2018). Essentials of Microbiology (em inglês). [S.l.]: Scientific e-Resources. p. 222-236. 348 páginas. ISBN 9781839473470
- ↑ McClintock, Barbara (1 de junho de 1950). «The origin and behavior of mutable loci in maize». Proceedings of the National Academy of Sciences (em inglês). 36 (6): 344–355. ISSN 0027-8424. PMC 1063197
 . PMID 15430309. doi:10.1073/pnas.36.6.344. Consultado em 25 de junho de 2020
. PMID 15430309. doi:10.1073/pnas.36.6.344. Consultado em 25 de junho de 2020
- ↑ Tsagris, Efthimia Mina; Alba, Ángel Emilio Martínez de; Gozmanova, Mariyana; Kalantidis, Kriton (2008). «Viroids». Cellular Microbiology (em inglês). 10 (11): 2168–2179. ISSN 1462-5822. PMID 18764915. doi:10.1111/j.1462-5822.2008.01231.x. Consultado em 25 de junho de 2020
- ↑ Mahy, B. W. J. (Brian W. J.); Van Regenmortel, M. H. V. (2010). Desk encyclopedia of general virology. Oxford, UK: Academic Press. p. 28. 644 páginas. ISBN 9780123751461. OCLC 480587729
- ↑ a b MADIGAN, M. T.; MARTINKO, J. M.; PARKER, J. (2003). Brock Biología de los Microorganismos. 10. [S.l.]: Madrid: Prentice-Hall. 1011 páginas. ISBN 9788420536798. OCLC 1105273048
- ↑ COLLIER, K. G. (abril de 1944). «THE ROLE OF PROJECTION IN THE GENESIS OF THE SUPER-EGO». British Journal of Medical Psychology. 20 (1): 96–99. ISSN 0007-1129. doi:10.1111/j.2044-8341.1944.tb00743.x. Consultado em 8 de junho de 2020
- ↑ Sandbulte, Matthew R.; Westgeest, Kim B.; Gao, Jin; Xu, Xiyan; Klimov, Alexander I.; Russell, Colin A.; Burke, David F.; Smith, Derek J.; Fouchier, Ron A. M. (20 de dezembro de 2011). «Discordant antigenic drift of neuraminidase and hemagglutinin in H1N1 and H3N2 influenza viruses». Proceedings of the National Academy of Sciences (em inglês). 108 (51): 20748–20753. ISSN 0027-8424. PMC 3251064
 . PMID 22143798. doi:10.1073/pnas.1113801108
. PMID 22143798. doi:10.1073/pnas.1113801108
- ↑ Moss, Ronald B.; Davey, Richard T.; Steigbigel, Roy T.; Fang, Fang (1 de junho de 2010). «Targeting pandemic influenza: a primer on influenza antivirals and drug resistance». Journal of Antimicrobial Chemotherapy (em inglês). 65 (6): 1086–1093. ISSN 0305-7453. doi:10.1093/jac/dkq100
- ↑ Hampson, Alan W.; Mackenzie, John S. (2006). «The influenza viruses». Medical Journal of Australia (em inglês). 185 (S10): S39–S43. ISSN 1326-5377. doi:10.5694/j.1326-5377.2006.tb00705.x
- ↑ Metzner, Karin J. (dezembro de 2006). «Detection and significance of minority quasispecies of drug-resistant HIV-1». Journal of HIV therapy. 11 (4): 74–81. ISSN 1462-0308. PMID 17578210
- ↑ a b c d e Câmara, Brunno (15 de setembro de 2013). «Morfologias virais». Biomedicina Padrão. Consultado em 27 de junho de 2020
- ↑ a b c d e f Roberto. O Quebra Cabeça Da Criação. [S.l.]: Clube de Autores (managed). p. 184-192. 465 páginas
- ↑ Shors, T (2008). Understanding Viruses (em inglês). Massachussetts: Jones & Bartlett Publishers. p. 73. 639 páginas. ISBN 978-0763729325
- ↑ «INTERNATIONAL COMMITTEE ON TAXONOMY OF VIRUSES». ICTVdb Descriptions. Consultado em 19 de abril de 2011. Arquivado do original em 19 de maio de 2011
- ↑ a b c d Sardinha dos Santos, Vanessa. «Vírus. Características gerais dos vírus». Mundo Educação. Consultado em 27 de junho de 2020
- ↑ Ferreira, Roberta Costa Santos; Riffel, Alessandro; Sant'Ana, Antônio Euzébio Goulart (2010). «HIV: mecanismo de replicação, alvos farmacológicos e inibição por produtos derivados de plantas». Química Nova. 33 (8): 1743–1755. ISSN 0100-4042. doi:10.1590/S0100-40422010000800023. Consultado em 26 de junho de 2020
- ↑ a b c Cruvinel, Wilson de Melo; Mesquita Júnior, Danilo; Araújo, Júlio Antônio Pereira; Catelan, Tânia Tieko Takao; Souza, Alexandre Wagner Silva de; Silva, Neusa Pereira da; Andrade, Luís Eduardo Coelho (agosto de 2010). «Sistema imunitário: Parte I. Fundamentos da imunidade inata com ênfase nos mecanismos moleculares e celulares da resposta inflamatória». Revista Brasileira de Reumatologia. 50 (4): 434–447. ISSN 0482-5004. doi:10.1590/S0482-50042010000400008. Consultado em 26 de junho de 2020
- ↑ a b Educacao, Portal. «Transcrição, tradução e replicação do Ácido Nucleico Viral». siteantigo.portaleducacao.com.br. Consultado em 13 de junho de 2020
- ↑ a b Simões, Rachel Siqueira de Queiroz (10 de dezembro de 2018). Virologia Humana e Veterinária. [S.l.]: Thieme Revinter Publicações LTDA. p. 314. ISBN 9788554650346
- ↑ Sardinha dos Santos, Vanessa. «Síntese proteica». Mundo Educação. Consultado em 12 de junho de 2020
- ↑ MURRAY, P. R.; ROSENTHAL, K. S.; PFAÜER, M. A.. Microbiología Médica. 5. ed. Madrid: Elsiver España, 2006. 976 p. ISBN 978-84-8174-927-4
- ↑ Ventura, Armando. «BMM 160 – Microbiologia Básica para Farmácia» (PDF). edisciplinas.usp.br. Consultado em 12 de junho de 2020
- ↑ Kirk da Fonseca Ribeiro, Krukemberghe Divino. «Molécula de RNA». Mundo Educação. Consultado em 12 de junho de 2020
- ↑ a b WAGNER, E. K.; HEWLETT, M. J.; BLOOM, D. C.; CAMERINI, D. (2007). Basic Virology. [S.l.]: Massachusetts: Wiley-Blackwell. p. 121-128. 580 páginas. ISBN 9781405147156. PMC 7152364
 . doi:10.1016/B978-0-323-07447-6.00013-2
. doi:10.1016/B978-0-323-07447-6.00013-2
- ↑ a b Komaroff AL. Is human herpesvirus-6 a trigger for chronic fatigue syndrome?. [S.l.]: Journal of Clinical Virology. p. S39–46. PMID 17276367. doi:10.1016/FS1386-6532(06)70010-5
- ↑ Komaroff, Anthony L. (1 de dezembro de 2006). «Is human herpesvirus-6 a trigger for chronic fatigue syndrome?». Journal of Clinical Virology. HHV-6: an underestimated virus (em inglês). 37: S39–S46. ISSN 1386-6532. doi:10.1016/S1386-6532(06)70010-5. Consultado em 8 de junho de 2020
- ↑ Fowler, Mary Glenn; Lampe, Margaret A.; Jamieson, Denise J.; Kourtis, Athena P.; Rogers, Martha F. (setembro de 2007). «Reducing the risk of mother-to-child human immunodeficiency virus transmission: past successes, current progress and challenges, and future directions». American Journal of Obstetrics and Gynecology. 197 (3): S3–S9. ISSN 0002-9378. doi:10.1016/j.ajog.2007.06.048
- ↑ Sauerbrei, Andreas; Wutzler, Peter (dezembro de 2000). «The Congenital Varicella Syndrome». Journal of Perinatology (em inglês). 20 (8): 548–554. ISSN 1476-5543. doi:10.1038/sj.jp.7200457
- ↑ Garnett, Geoffrey P. (1 de fevereiro de 2005). «Role of Herd Immunity in Determining the Effect of Vaccines against Sexually Transmitted Disease». The Journal of Infectious Diseases (em inglês). 191 (Supplement_1): S97–S106. ISSN 0022-1899. doi:10.1086/425271. Consultado em 8 de junho de 2020
- ↑ Platonov, A. E. (2006). «The influence of weather conditions on the epidemiology of vector-borne diseases by the example of West Nile fever in Russia». Vestnik Rossiiskoi Akademii Meditsinskikh Nauk (2): 25–29. ISSN 0869-6047. PMID 16544901. Consultado em 8 de junho de 2020
- ↑ Common Viral Infections (em inglês). [S.l.]: PediaPress. p. 38. 387 páginas
- ↑ Collier, J. (27 de maio de 1882). «Meinardus Schotanus». Notes and Queries. s6–V (126): 409–409. ISSN 1471-6941. doi:10.1093/nq/s6-v.126.409a
- ↑ Patterson, K. D.; Pyle, G. F. (1991). «The geography and mortality of the 1918 influenza pandemic». Bulletin of the History of Medicine. 65 (1): 4–21. ISSN 0007-5140. PMID 2021692
- ↑ Johnson, Niall P. A. S.; Mueller, Juergen (1 de março de 2002). «Updating the Accounts: Global Mortality of the 1918-1920 "Spanish" Influenza Pandemic». Bulletin of the History of Medicine (em inglês). 76 (1): 105–115. ISSN 1086-3176. doi:10.1353/bhm.2002.0022
- ↑ Gabriel, W. B. (20 de setembro de 1952). «Cancer of the Rectum». British Medical Journal. 2 (4785): 653–654. ISSN 0007-1447. PMC 2021692

- ↑ Eisinger, Robert Walter; Fauci, Anthony S. «Ending the HIV/AIDS Pandemic - Volume 24, Number 3—March 2018 - Emerging Infectious Diseases journal - CDC» (em inglês). 24 (3): 413–16. ISSN 1080-6059. PMC 5823353
 . PMID 29460740. doi:10.3201/eid2403.171797. Consultado em 8 de junho de 2020
. PMID 29460740. doi:10.3201/eid2403.171797. Consultado em 8 de junho de 2020
- ↑ Qin, Y.; Zhao, M. J.; Tan, Y. Y.; Li, X. Q.; Zheng, J. D.; Peng, Z. B.; Feng, L. Z. (10 de agosto de 2018). «History of influenza pandemics in China during the past century». Zhonghua Liu Xing Bing Xue Za Zhi = Zhonghua Liuxingbingxue Zazhi. 39 (8): 1028–1031. ISSN 0254-6450. PMID 30180422. doi:10.3760/cma.j.issn.0254-6450.2018.08.003
- ↑ Gao, Feng; Bailes, Elizabeth; Robertson, David L.; Chen, Yalu; Rodenburg, Cynthia M.; Michael, Scott F.; Cummins, Larry B.; Arthur, Larry O.; Peeters, Martine (fevereiro de 1999). «Origin of HIV-1 in the chimpanzee Pan troglodytes troglodytes». Nature (em inglês). 397 (6718): 436–441. ISSN 1476-4687. doi:10.1038/17130
- ↑ Mawar, Nita; Saha, Seema; Pandit, Apoorvaa; Mahajan, Uma (dezembro de 2005). «The third phase of HIV pandemic: social consequences of HIV/AIDS stigma & discrimination & future needs». The Indian Journal of Medical Research. 122 (6): 471–484. ISSN 0971-5916. PMID 16517997. Consultado em 8 de junho de 2020
- ↑ «Status of the global HIV epidemic» (PDF). data.unaids.org. 2008. Consultado em 8 de junho de 2020. Arquivado do original (PDF) em 22 de novembro de 2015
- ↑ Towner, Jonathan S.; Khristova, Marina L.; Sealy, Tara K.; Vincent, Martin J.; Erickson, Bobbie R.; Bawiec, Darcy A.; Hartman, Amy L.; Comer, James A.; Zaki, Sherif R. (1 de julho de 2006). «Marburgvirus Genomics and Association with a Large Hemorrhagic Fever Outbreak in Angola». Journal of Virology (em inglês). 80 (13): 6497–6516. ISSN 0022-538X. PMC 1488971
 . PMID 16775337. doi:10.1128/JVI.00069-06. Consultado em 25 de junho de 2020
. PMID 16775337. doi:10.1128/JVI.00069-06. Consultado em 25 de junho de 2020
- ↑ «World Health Organisation report, 24 September 2014» (PDF). Organização Mundial da Saúde. 24 de setembro de 2014. Consultado em 25 de junho de 2020
- ↑ Weiss, Susan R.; Leibowitz, Julian L. (1 de janeiro de 2011). Maramorosch, Karl; Shatkin, Aaron J.; Murphy, Frederick A., eds. «Chapter 4 - Coronavirus Pathogenesis». Academic Press (em inglês). 81: 85–164. PMC 7149603
 . PMID 22094080. doi:10.1016/b978-0-12-385885-6.00009-2
. PMID 22094080. doi:10.1016/b978-0-12-385885-6.00009-2
- ↑ Wong, Andrew T. Y.; Chen, Hong; Liu, Shao-haei; Hsu, Enoch K.; Luk, Kristine S.; Lai, Christopher K. C.; Chan, Regina F. Y.; Tsang, Owen T. Y.; Choi, K. W. (15 de maio de 2017). «From SARS to Avian Influenza Preparedness in Hong Kong». Clinical Infectious Diseases (em inglês). 64 (suppl_2): S98–S104. ISSN 1058-4838. doi:10.1093/cid/cix123
- ↑ Ashour, Hossam M.; Elkhatib, Walid F.; Rahman, Md Masudur; Elshabrawy, Hatem A. (março de 2020). «Insights into the Recent 2019 Novel Coronavirus (SARS-CoV-2) in Light of Past Human Coronavirus Outbreaks». Pathogens (em inglês). 9 (3): 186. PMC 7157630
 . PMID 32143502. doi:10.3390/pathogens9030186. Consultado em 25 de junho de 2020
. PMID 32143502. doi:10.3390/pathogens9030186. Consultado em 25 de junho de 2020
- ↑ Deng, Sheng-Qun; Peng, Hong-Juan (fevereiro de 2020). «Characteristics of and Public Health Responses to the Coronavirus Disease 2019 Outbreak in China». Journal of Clinical Medicine (em inglês). 9 (2): 575. PMC 7074453
 . PMID 32093211. doi:10.3390/jcm9020575. Consultado em 25 de junho de 2020
. PMID 32093211. doi:10.3390/jcm9020575. Consultado em 25 de junho de 2020
- ↑ Salcedo, Andrea; Yar, Sanam; Cherelus, Gina (8 de maio de 2020). «Coronavirus Travel Restrictions, Across the Globe». The New York Times. Consultado em 25 de junho de 2020
- ↑ Einstein, Mark H; Schiller, John T; Viscidi, Raphael P; Strickler, Howard D; Coursaget, Pierre; Tan, Tina; Halsey, Neal; Jenkins, David (junho de 2009). «Clinician's guide to human papillomavirus immunology: knowns and unknowns». The Lancet Infectious Diseases. 9 (6): 347–356. ISSN 1473-3099. doi:10.1016/s1473-3099(09)70108-2. Consultado em 8 de junho de 2020
- ↑ Shuda, M.; Feng, H.; Kwun, H. J.; Rosen, S. T.; Gjoerup, O.; Moore, P. S.; Chang, Y. (23 de setembro de 2008). «T antigen mutations are a human tumor-specific signature for Merkel cell polyomavirus». Proceedings of the National Academy of Sciences (em inglês). 105 (42): 16272–16277. ISSN 0027-8424. PMC 2551627
 . PMID 18812503. doi:10.1073/pnas.0806526105. Consultado em 8 de junho de 2020
. PMID 18812503. doi:10.1073/pnas.0806526105. Consultado em 8 de junho de 2020
- ↑ Leão, J. C.; Hinrichsen, S. L.; Freitas, B. L. de; Porter, S. R. (março de 1999). «Herpes vírus humano-8 e Sarcoma de Kaposi». Revista da Associação Médica Brasileira. 45 (1): 55–62. ISSN 0104-4230. doi:10.1590/S0104-42301999000100011
- ↑ Pulitzer, Melissa P.; Amin, Bijal D.; Busam, Klaus J. (maio de 2009). «Merkel Cell Carcinoma: Review». Advances in Anatomic Pathology (em inglês). 16 (3): 135–144. ISSN 1072-4109. doi:10.1097/PAP.0b013e3181a12f5a
- ↑ Schatzmayr, Hermann G. (2003). «Novas perspectivas em vacinas virais». História, Ciências, Saúde-Manguinhos. 10: 655–669. ISSN 0104-5970. doi:10.1590/S0104-59702003000500010. Consultado em 12 de junho de 2020
- ↑ Campos, André Luiz Vieira de; Nascimento, Dilene Raimundo do; Maranhão, Eduardo (2003). «A história da poliomielite no Brasil e seu controle por imunização». História, Ciências, Saúde-Manguinhos. 10: 573–600. ISSN 0104-5970. doi:10.1590/S0104-59702003000500007
- ↑ KNIPE, D. M.; HOWLEY, P. M.; GRIFFIN, D. E.; LAMB, R. A.; MARTIN, M. A.; ROIZMAN, B.; STRAUS, S. E.. Fields - Virology. 4. ed. Philadelphia: Lippincott Williams & Wilkins, 2001. 3280 p. ISBN 978-0-7817-1832-5
- ↑ Santos, Neusa de Queiroz (2004). «A resistência bacteriana no contexto da infecção hospitalar». Texto & Contexto - Enfermagem. 13 (SPE): 64–70. ISSN 0104-0707. doi:10.1590/S0104-07072004000500007. Consultado em 27 de junho de 2020
- ↑ La Scola, Bernard; Desnues, Christelle; Pagnier, Isabelle; Robert, Catherine; Barrassi, Lina; Fournous, Ghislain; Merchat, Michèle; Suzan-Monti, Marie; Forterre, Patrick (setembro de 2008). «The virophage as a unique parasite of the giant mimivirus». Nature (em inglês). 455 (7209): 100–104. ISSN 1476-4687. doi:10.1038/nature07218
- ↑ Nickel, Osmar; Martins Fajardo, Thor Vinícius (março de 2019). «Transmissão de vírus e controle de viroses em plantas» (PDF). ainfo.cnptia.embrapa.br. ISSN 1808-4648. Consultado em 13 de junho de 2020
- ↑ Araguaia, Mariana. «Vírus de planta». Brasil Escola. Consultado em 13 de junho de 2020
- ↑ Shors, Tracey J. (1994). «A Stress-Induced Facilitation of Associative Learning». PsycEXTRA Dataset. Consultado em 13 de junho de 2020
- ↑ «BIBLIOGRAPHY». Elsevier. 1967: 799–807. ISBN 978-1-4831-9761-6
- ↑ a b Wommack, K. Eric; Colwell, Rita R. (1 de março de 2000). «Virioplankton: Viruses in Aquatic Ecosystems». Microbiology and Molecular Biology Reviews (em inglês). 64 (1): 69–114. ISSN 1092-2172. PMC 98987
 . PMID 10704475. doi:10.1128/MMBR.64.1.69-114.2000. Consultado em 13 de junho de 2020
. PMID 10704475. doi:10.1128/MMBR.64.1.69-114.2000. Consultado em 13 de junho de 2020
- ↑ Bergh, Øivind; BØrsheim, Knut Yngve; Bratbak, Gunnar; Heldal, Mikal (agosto de 1989). «High abundance of viruses found in aquatic environments». Nature (em inglês). 340 (6233): 467–468. ISSN 1476-4687. doi:10.1038/340467a0
- ↑ Shors, Stephanie; Miller, Frank H. (2010). «Case 393». Elsevier: 834–835. ISBN 978-1-4160-5944-8. Consultado em 13 de junho de 2020
- ↑ Goris, Nesya; Vandenbussche, Frank; De Clercq, Kris (1 de abril de 2008). «Potential of antiviral therapy and prophylaxis for controlling RNA viral infections of livestock». Antiviral Research. Special Issue: Treatment of highly pathogenic RNA viral infections (em inglês). 78 (1): 170–178. ISSN 0166-3542. doi:10.1016/j.antiviral.2007.10.003
- ↑ Carmichael, L. E. (2005). «An Annotated Historical Account of Canine Parvovirus». Journal of Veterinary Medicine, Series B (em inglês). 52 (7-8): 303–311. ISSN 1439-0450. doi:10.1111/j.1439-0450.2005.00868.x
- ↑ Chen, Yanping; Zhao, Yan; Hammond, John; Hsu, Hei-ti; Evans, Jay; Feldlaufer, Mark (1 de outubro de 2004). «Multiple virus infections in the honey bee and genome divergence of honey bee viruses». Journal of Invertebrate Pathology (em inglês). 87 (2): 84–93. ISSN 0022-2011. doi:10.1016/j.jip.2004.07.005
- ↑ Prangishvili, David; Forterre, Patrick; Garrett, Roger A. (novembro de 2006). «Viruses of the Archaea: a unifying view». Nature Reviews Microbiology (em inglês). 4 (11): 837–848. ISSN 1740-1534. doi:10.1038/nrmicro1527. Consultado em 13 de junho de 2020
- ↑ Prangishvili, D.; Garrett, R. A. (1 de abril de 2004). «Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses». Biochemical Society Transactions (em inglês). 32 (2): 204–208. ISSN 0300-5127. doi:10.1042/bst0320204
- ↑ Mojica, Francisco J.M.; Díez-Villaseñor, Chc)sar; García-Martínez, Jesús; Soria, Elena (1 de fevereiro de 2005). «Intervening Sequences of Regularly Spaced Prokaryotic Repeats Derive from Foreign Genetic Elements». Journal of Molecular Evolution (em inglês). 60 (2): 174–182. ISSN 1432-1432. doi:10.1007/s00239-004-0046-3
- ↑ Makarova, Kira S.; Grishin, Nick V.; Shabalina, Svetlana A.; Wolf, Yuri I.; Koonin, Eugene V. (16 de março de 2006). «A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action». Biology Direct. 1 (1): 7. ISSN 1745-6150. PMC 1462988
 . PMID 16545108. doi:10.1186/1745-6150-1-7
. PMID 16545108. doi:10.1186/1745-6150-1-7
- ↑ van der Oost, John; Westra, Edze R.; Jackson, Ryan N.; Wiedenheft, Blake (julho de 2014). «Unravelling the structural and mechanistic basis of CRISPR–Cas systems». Nature Reviews Microbiology (em inglês). 12 (7): 479–492. ISSN 1740-1534. PMC 4225775
 . PMID 24909109. doi:10.1038/nrmicro3279. Consultado em 13 de junho de 2020
. PMID 24909109. doi:10.1038/nrmicro3279. Consultado em 13 de junho de 2020
- ↑ Dávila-Ramos, Sonia; Castelán-Sánchez, Hugo G.; Martínez-Ávila, Liliana; Sánchez-Carbente, María del Rayo; Peralta, Raúl; Hernández-Mendoza, Armando; Dobson, Alan D. W.; Gonzalez, Ramón A.; Pastor, Nina (2019). «A Review on Viral Metagenomics in Extreme Environments». Frontiers in Microbiology (em inglês). 10: 2403. ISSN 1664-302X. PMC 6842933
 . PMID 31749771. doi:10.3389/fmicb.2019.02403. Consultado em 13 de junho de 2020
. PMID 31749771. doi:10.3389/fmicb.2019.02403. Consultado em 13 de junho de 2020
- ↑ Zhang, Qi-Ya; Gui, Jian-Fang (1 de dezembro de 2018). «Diversity, evolutionary contribution and ecological roles of aquatic viruses». Science China Life Sciences (em inglês). 61 (12): 1486–1502. ISSN 1869-1889. doi:10.1007/s11427-018-9414-7. Consultado em 13 de junho de 2020
- ↑ Suttle, Curtis A. (setembro de 2005). «Viruses in the sea». Nature (em inglês). 437 (7057): 356–361. ISSN 1476-4687. doi:10.1038/nature04160. Consultado em 13 de junho de 2020
- ↑ Wilhelm, Steven W.; Suttle, Curtis A. (1 de outubro de 1999). «Viruses and Nutrient Cycles in the SeaViruses play critical roles in the structure and function of aquatic food webs». BioScience (em inglês). 49 (10): 781–788. ISSN 0006-3568. doi:10.2307/1313569. Consultado em 13 de junho de 2020
- ↑ Shelford, Emma J.; Suttle, Curtis A. (9 de fevereiro de 2018). «Virus-mediated transfer of nitrogen from heterotrophic bacteria to phytoplankton». Biogeosciences (em inglês). 15 (3): 809–819. ISSN 1726-4170. doi:10.5194/bg-15-809-2018. Consultado em 13 de junho de 2020
- ↑ Wigington, Charles H.; Sonderegger, Derek; Brussaard, Corina P. D.; Buchan, Alison; Finke, Jan F.; Fuhrman, Jed A.; Lennon, Jay T.; Middelboe, Mathias; Suttle, Curtis A. (25 de janeiro de 2016). «Re-examination of the relationship between marine virus and microbial cell abundances». Nature Microbiology (em inglês). 1 (3): 1–9. ISSN 2058-5276. doi:10.1038/nmicrobiol.2015.24. Consultado em 13 de junho de 2020
- ↑ Brussaard, Corina P. D. (2004). «Viral Control of Phytoplankton Populations—a Review1». Journal of Eukaryotic Microbiology (em inglês). 51 (2): 125–138. ISSN 1550-7408. doi:10.1111/j.1550-7408.2004.tb00537.x. Consultado em 13 de junho de 2020
- ↑ Suttle, Curtis A. (outubro de 2007). «Marine viruses — major players in the global ecosystem». Nature Reviews Microbiology (em inglês). 5 (10): 801–812. ISSN 1740-1534. doi:10.1038/nrmicro1750. Consultado em 13 de junho de 2020
- ↑ Cunico, Wilson; Gomes, Claudia R. B.; Vellasco Junior, Walcimar T. (2008). «HIV - recentes avanços na pesquisa de fármacos». Química Nova. 31 (8): 2111–2117. ISSN 0100-4042. doi:10.1590/S0100-40422008000800035. Consultado em 12 de junho de 2020
- ↑ Moraes, Paula Louredo. «Características gerais dos vírus». Brasil Escola. Consultado em 27 de junho de 2020
- ↑ a b c d e Eiras, Marcelo; Daròs, Jose Antonio; Flores, Ricardo; Kitajima, Elliot W. (junho de 2006). «Viróides e virusóides: relíquias do mundo de RNA». Fitopatologia Brasileira. 31 (3): 229–246. ISSN 0100-4158. doi:10.1590/S0100-41582006000300001
- ↑ a b Korsman, Stephen N. J.; Zyl, Gert Van; Nutt, Louise; Andersson, Monique I.; Preiser, Wolfgang (10 de dezembro de 2014). Virologia. [S.l.]: Elsevier Brasil. ISBN 9788535279771
- ↑ Brites, Alice Dantas. «Príons: Proteínas anormais provocam doenças». educacao.uol.com.br. Consultado em 27 de junho de 2020
- ↑ Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James (2000). «Viruses: Structure, Function, and Uses». Molecular Cell Biology. 4th edition (em inglês) (6): 3. Consultado em 13 de junho de 2020
- ↑ Collier, R. J.; Collier, J. L., eds. (7 de janeiro de 2012). «Environmental Physiology of Livestock». doi:10.1002/9781119949091. Consultado em 13 de junho de 2020
- ↑ Matsuzaki, Shigenobu; Rashel, Mohammad; Uchiyama, Jumpei; Sakurai, Shingo; Ujihara, Takako; Kuroda, Masayuki; Imai, Shosuke; Ikeuchi, Masahiko; Tani, Toshikazu (2005). «Bacteriophage therapy: a revitalized therapy against bacterial infectious diseases». Journal of Infection and Chemotherapy. 11 (5): 211–219. ISSN 1341-321X. doi:10.1007/s10156-005-0408-9. Consultado em 13 de junho de 2020
- ↑ a b ZILINSKAS, R.A. (1 de agosto de 2017). «A brief history of biological weapons programmes and the use of animal pathogens as biological warfare agents». Revue Scientifique et Technique de l'OIE. 36 (2): 415–422. ISSN 0253-1933. doi:10.20506/rst.36.2.2662. Consultado em 13 de junho de 2020
- ↑ a b Artenstein, Andrew W.; Grabenstein, John D. (1 de outubro de 2008). «Smallpox vaccines for biodefense: need and feasibility». Expert Review of Vaccines. 7 (8): 1225–1237. ISSN 1476-0584. doi:10.1586/14760584.7.8.1225. Consultado em 13 de junho de 2020
- ↑ Ngo-Duc, Tam-Triet; Plank, Joshua M.; Chen, Gongde; Harrison, Reed E. S.; Morikis, Dimitrios; Liu, Haizhou; Haberer, Elaine D. (2018). «M13 bacteriophage spheroids as scaffolds for directed synthesis of spiky gold nanostructures». Nanoscale (em inglês). 10 (27): 13055–13063. ISSN 2040-3364. doi:10.1039/C8NR03229G. Consultado em 13 de junho de 2020
- ↑ a b Fischlechner, Martin; Donath, Edwin (2007). «Viruses as Building Blocks for Materials and Devices». Angewandte Chemie International Edition (em inglês). 46 (18): 3184–3193. ISSN 1521-3773. doi:10.1002/anie.200603445. Consultado em 13 de junho de 2020
- ↑ Soto, Carissa M.; Blum, Amy Szuchmacher; Vora, Gary J.; Lebedev, Nikolai; Meador, Carolyn E.; Won, Angela P.; Chatterji, Anju; Johnson, John E.; Ratna, Banahalli R. (1 de abril de 2006). «Fluorescent Signal Amplification of Carbocyanine Dyes Using Engineered Viral Nanoparticles». Journal of the American Chemical Society. 128 (15): 5184–5189. ISSN 0002-7863. doi:10.1021/ja058574x. Consultado em 13 de junho de 2020
- ↑ Blum, Amy Szuchmacher; Soto, Carissa M.; Wilson, Charmaine D.; Brower, Tina L.; Pollack, Steven K.; Schull, Terence L.; Chatterji, Anju; Lin, Tianwei; Johnson, John E. (julho de 2005). «An Engineered Virus as a Scaffold for Three-Dimensional Self-Assembly on the Nanoscale». Small (em inglês). 1 (7): 702–706. ISSN 1613-6810. doi:10.1002/smll.200500021. Consultado em 13 de junho de 2020
- ↑ Cello, Jeronimo; Paul, Aniko V.; Wimmer, Eckard (9 de agosto de 2002). «Chemical Synthesis of Poliovirus cDNA: Generation of Infectious Virus in the Absence of Natural Template». Science (em inglês). 297 (5583): 1016–1018. ISSN 0036-8075. PMID 12114528. doi:10.1126/science.1072266. Consultado em 13 de junho de 2020
- ↑ a b Coleman, J. Robert; Papamichail, Dimitris; Skiena, Steven; Futcher, Bruce; Wimmer, Eckard; Mueller, Steffen (27 de junho de 2008). «Virus Attenuation by Genome-Scale Changes in Codon Pair Bias». Science (em inglês). 320 (5884): 1784–1787. ISSN 0036-8075. PMC 2754401
 . PMID 18583614. doi:10.1126/science.1155761. Consultado em 13 de junho de 2020
. PMID 18583614. doi:10.1126/science.1155761. Consultado em 13 de junho de 2020
Bibliografia
[editar | editar código-fonte]- Iwanowski, D. (1892). Über die Mosaikkrankheit der Tabakspflanze. Bulletin Scientifique publié par l'Académie Impériale des Sciences de Saint-Pétersbourg / Nouvelle Serie III. 35. [S.l.: s.n.]
- Collier, Leslie; Balows, Albert; Sussman, Max (1998). Topley and Wilson's Microbiology and Microbial Infections. 1. [S.l.: s.n.] ISBN 0340663162
- Beijerinck, M.W. (1898). Über ein Contagium vivum fluidum als Ursache der Fleckenkrankheit der Tabaksblätter. Verhandelingen der Koninklyke akademie van Wettenschappen te Amsterdam. 65. [S.l.: s.n.]
- Horzinek, Marian C. (1997). The birth of virology. Antonie van Leeuwenhoek. 71. [S.l.: s.n.] PMID 9049014. doi:10.1023/A:1000197505492
- Zaitlin, Milton (1998). «The Discovery of the Causal Agent of the Tobacco Mosaic Disease» (PDF). In: Kung, S. D.; Yang, S. F. Discoveries in Plant Biology. Hong Kong: World Publishing Co. ISBN 9789810213138
- Vieira, Francisco Cleber Sousa; Nahas, Ely (setembro de 2000). Quantificação de bactérias totais e esporuladas no solo. Scientia Agricola. 57. [S.l.: s.n.] ISSN 0103-9016. doi:10.1590/S0103-90162000000300026
- Rosen, Fred S. (2004). Isolation of Poliovirus — John Enders and the Nobel Prize. New England Journal of Medicine. 351. [S.l.: s.n.] ISSN 0028-4793. PMID 15470207. doi:10.1056/NEJMp048202
- Bawden, F.C.; Pirie, N.W. (1937). The isolation and some properties of liquid crystalline substances from solanaceous plants infected with threestrains of tobacco mosaic virus. Proc R Soc Med. 123. [S.l.: s.n.]







