Retroviridae
| Realm | Riboviria |
|---|---|
| Règne | Pararnavirae |
| Embranchement | Artverviricota |
| Classe | Revtraviricetes |
| Ordre | Ortervirales |
Genres de rang inférieur
Les Retroviridae (rétrovirus) sont une famille de virus qui regroupe les sous-familles suivantes : Orthoretrovirinae (en) et Spumaretrovirinae (en). Ce sont des virus à ARN monocaténaire de polarité positive infectant les vertébrés. Ils se distinguent notamment par la présence d'une enzyme virale : la transcriptase inverse (TI, ou encore RT pour reverse transcriptase), qui rétrotranscrit leur génome d'ARN en ADN pour être intégré par la suite dans le génome de la cellule hôte. La TI a la particularité de commettre assez facilement des erreurs, ce qui fait que certains rétrovirus ont une grande variabilité génétique. Les Retroviridae disposent d'un fort pouvoir oncogène.
Le virus de l'immunodéficience humaine (VIH), responsable du Sida, est un rétrovirus. À cause de sa grande variabilité, il est très compliqué pour les chercheurs d'élaborer un vaccin, car les glycoprotéines transmembranaires de l'enveloppe du virus (GP120 et GP41) mutent constamment.
Cycle de vie d'un rétrovirus endogène:
|
Morphologie
[modifier | modifier le code]Ce sont des virus enveloppés, de 110 à 125 nanomètres. Leur enveloppe est issue de la dernière cellule infectée, car la prolifération se fait par bourgeonnement. Elle est enrichie par des protéines d'enveloppes spécifiques codées par le gène env du virus. Autour de l'ARN se trouve la capside. Le génome est diploïde, les deux brins monocaténaires d'ARN sont reliés par des ponts hydrogène à leur extrémité 5'.
Pour pouvoir s'intégrer dans le génome de la cellule hôte et synthétiser ses protéines virales via la machinerie cellulaire, qui est bicaténaire, la transcriptase inverse va synthétiser les deuxièmes brins manquants pour donner un ADN et non plus un ARN. Le brin d'ARN étant monocaténaire, des erreurs de transcription surviennent fréquemment (il n'y a pas de contrôle possible à l'aide du nucléotide complémentaire) ; si certaines aboutissent à un ADN improductif, d'autres sont viables et engendrent des mutants qui peuvent éventuellement différer par leur signature antigénique. Cette grande variabilité rend difficile la vaccination.
Organisation du génome
[modifier | modifier le code]Le génome se décompose en différentes régions, ayant chacune un rôle bien défini. Orienté de 5' vers 3' :
- la séquence R : il s'agit d'une séquence répétée qui sert à former la première fibre d'ADN négative en permettant le premier saut réplicatif ;
- U5 : une région unique qui joue un rôle dans la terminaison de la synthèse d'ARN viral ;
- séquence Leader : elle contient 2 parties :
- SD : le site donneur d'épissage pour l'ARN subgénomique,
- Ψ : le signal d'empaquetage de l'ARN génomique, c'est-à-dire l'encapsidation ;
- PBS : pour Primer Binding Site, c'est le site d'attache de l'amorce pour la synthèse du brin d'ADN complémentaire par la TI. Le PBS est associé à un ARNt.
Puis, suivent les trois gènes de structure :
- gag : (pour group specific antigens ) ce gène code la polyprotéine structurale du virus, contenant les domaines de matrice (MA), capside(CA) et nucleocapside (NC) ;
- pol : (pour polymérase) ce gène code la transcriptase inverse, l'intégrase qui permet l'intégration d'ADN bicaténaire viral dans l'ADN cellulaire, une protéase qui clive les domaines de gag et pol lors de la maturation, une endonucléase et une RNase H ;
- env : (pour enveloppe) ce gène code des protéines d'enveloppe, les spicules.
Enfin, la dernière région :
- PPT : pour PolyPurine Tract sert d'amorce et empêche la digestion par la Transcriptase Inverse
- U3 : contient le promoteur et l'enhancer pour la transcription du génome, ainsi que le signal de polyadénylation de l'ARN ;
- séquence R.

Rétroéléments
[modifier | modifier le code]Ils sont l'un des moteurs de la biodiversité, par leurs propriétés de transposition[2]. Les rétrovirus sont un cas particulier, avec une « transposition » qui propage dans ce cas une infection à partir de la cellule pénétrée par le virus aux cellules et organes voisins[2].
Un rétrotransposon peut être converti en agent infectieux par l'acquisition d'une glycoprotéine d'enveloppe (« capture d’enveloppe »), ce qui a permis l'apparition de rétrovirus infectieux de mammifères[2]. Ces captures expliqueraient les parentés étroites de protéines Env (protéines d'enveloppe) de différents rétrovirus pourtant phylogéniquement distants. Comprendre ces « étymologies » rétrovirales est nécessaire pour l'établissement de la phylogénie des rétrovirus et la physiopathologie des infections qu'ils produisent, et peut-être pour la découverte de nouveaux rétroéléments[2].
Oncogenèse
[modifier | modifier le code]Certains rétrovirus possèdent, en plus, un oncogène. Cet oncogène code une protéine de transformation. Cette protéine confère aux virus un fort pouvoir de transformation leur permettant d'induire des tumeurs très rapidement (quelques semaines) après l'infection. Les protéines oncogéniques peuvent être de différentes natures :
- protéines impliquées dans l’activité enzymatique : pp60 src (RSV), c'est une protéine kinase membranaire régulant le fonctionnement de nombreuses protéines ;
- analogues de facteurs de croissance cellulaire ou de leurs récepteurs :
- Sis : analogue muté du PDGF, Sis se fixe sur le récepteur du PDGF,
- Erb : analogue muté du récepteur à l’EGF ;
- protéines fixant la GTP : Ras (active la voie des MAP Kinases) ;
- protéines à activité nucléaire : Myc, Myb, Fos, sont des facteurs de transcription impliqués dans la prolifération cellulaire.
Classification
[modifier | modifier le code]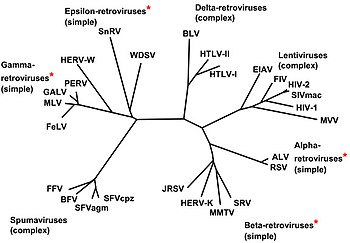
On classe les rétrovirus en deux grandes catégories :
- les rétrovirus exogènes, qui ne sont pas présents naturellement dans l'organisme et qui ont donc besoin de l'infecter pour effectuer leur cycle de réplication et ainsi produire des virions ;
- les rétrovirus endogènes, dont le matériel génétique est présent au sein même du génome de l'organisme, mais qui n'est généralement pas exprimé.
Exogène
[modifier | modifier le code]Seuls les rétrovirus exogènes sont formellement classifiés par l'International Committee on Taxonomy of Viruses (ICTV)[3] et sont regroupés dans deux sous-familles :
- orthoretrovirinae ;
- spumaretrovirinae.
Ces deux sous-familles comprennent au total sept genres : Alpharetrovirus, Betaretrovirus, Gammaretrovirus, Deltaretrovirus, Epsilonretrovirus, Lentivirus et Spumavirus.
Selon leur aspect infectieux, les rétrovirus sont classés dans trois catégories :
- les cinq genres de rétrovirus les plus anciennement connus (tous ceux appartenant à la sous-famille orthoretrovirinae à l'exception des lentivirus) induisent des leucémies et des cancers chez les organismes infectés, ils sont oncogènes et on les appelle également des oncovirus ;
- les lentivirus , dont fait partie le VIH, détruisent généralement les cellules qu'elles infectent, ils sont cytopathogènes ;
- pour l'instant on ne connaît aucune maladie induite par les spumavirus.
Endogène
[modifier | modifier le code]L'ADN de toutes les espèces, mammifères y compris, comporte des génomes viraux laissés par des virus, probablement à l'occasion d'une infection de cellules germinales, puis dupliqués avec l'ADN de génération en génération. Ces rétrovirus endogènes peuvent être présents en très grand nombre dans les génomes, où ils sont présents en de multiples exemplaires. Ils appartiennent à ce qu'on appelle les répétitions dispersées. Le génome humain contient environ 450 000 exemplaires de rétrovirus endogènes, plus ou moins délétés ou mutés depuis leur insertion dans la lignée germinale humaine[4].
L'expression des gènes des rétrovirus endogènes est normalement inhibée par une protéine spécialisée dite « KAP1 », dès le début de l'embryogenèse[5]. L'étude des rétrovirus endogènes est un enjeu médical, car ils peuvent être source de diverses maladies (dont des cancers) quand leur génome est exprimé, malgré les protections naturelles mises en place au cours de l'évolution.
Histoire
[modifier | modifier le code]Dès le début, le caractère oncogène des rétrovirus a été observé. C'est ainsi qu'en 1908, le virus de la leucémie aviaire (ALV) a été transmis d'un poulet à un autre par les Danois Vilhelm Ellerman et Oluf Bang après transfert de tissus infectés. En 1911, c'est au tour de Peyton Rous de mettre en évidence le virus du sarcome de Rous (RSV), qui, contrairement à l'ALV, induit une tumeur quelques semaines après l'infection, contre plusieurs mois pour l'ALV[6].
Mais il faudra attendre plus de quarante ans pour observer un rétrovirus chez les mammifères, avec la découverte du virus de la leucémie murine (MLV) en 1957 par Ludwig Gross et enfin 1981 pour la découverte du HTLV-1 par Robert Gallo, faisant de ce virus le premier rétrovirus humain identifié. Très rapidement d'autres virus sont identifiés : HTLV-2 en 1982 et surtout le VIH en 1983.
La découverte de ce dernier et la pandémie que l'on connaît depuis ont poussé les institutions de recherche publiques et privées, ainsi que l'industrie pharmaceutique, à faire des rétrovirus les virus les plus étudiés au monde. Une nouvelle classe d'antiviraux a été mise au point, pour s'attaquer à des particularités des rétrovirus, et regroupe ainsi ceux appelés antirétroviraux.
Perspective pour la recherche scientifique
[modifier | modifier le code]Des manipulations génétiques ont montré que l'on pouvait utiliser des rétrovirus pour amener des gènes particuliers dans une cellule humaine. Les chercheurs espèrent ainsi introduire dans l'ADN de personnes atteintes de déficiences génétiques héréditaires les gènes qui leur font défaut. On pourrait ainsi infecter positivement un organisme humain et le guérir de maladies génétiques. Une maîtrise totale de ces techniques pourrait à long terme permettre de réintroduire dans le génome humain les gènes déficients communs à l'espèce humaine, comme ceux permettant de synthétiser certaines vitamines (vitamine C par exemple), les acides aminés essentiels et les acides gras essentiels. Ces nutriments doivent actuellement être obligatoirement apportés par la nourriture.[réf. nécessaire]
Il y a sans doute une question bioéthique de fond pour réguler ces manipulations.[réf. nécessaire]
Notes et références
[modifier | modifier le code]- ICTV. International Committee on Taxonomy of Viruses. Taxonomy history. Published on the Internet https://talk.ictvonline.org/., consulté le 25 janvier 2021
- (en) John M. Coffin, Stephen H. Hughes et Harold E. Varmus, « Structural Classes of Retroelements and Replication Strategies », dans Retroviruses, Cold Spring Harbor Laboratory Press, (ISBN 0-87969-571-4, lire en ligne)
- (en) Family : Retroviridae, selon le International Committee on Taxonomy of Viruses.
- (en) International Human Genome Sequencing Consortium, « Initial sequencing and analysis of the human genome. », Nature, vol. 409, , p. 820-921 (PMID 11237011).
- Helen M. Rowe, Johan Jakobsson, Daniel Mesnard, Jacques Rougemont, Séverine Reynard, Tugce Aktas, Pierre V. Maillard, Hillary Layard-Liesching, Sonia Verp, Julien Marquis, François Spitz, Daniel B. Constam & Didier Trono ; KAP1 controls endogenous retroviruses in embryonic stem cells ; Nature 463, 237-240 ; 14 janvier 2010 ; doi:10.1038/nature08674.
- Sous la direction de Jean-Marie Huraux, Traité de virologie médicale, Paris, Éditions Estem, , 699 p. (ISBN 2-84371-203-3), p. 83.
Références biologiques
[modifier | modifier le code]- (en) Référence ICTV : Retroviridae (consulté le )
Bibliographie
[modifier | modifier le code]- (en) Retroviruses, sur le site du National Center for Biotechnology Information (NCBI)
- (en) John M. Coffin, Stephen H. Hughes et Harold E. Varmus, « Structural Classes of Retroelements and Replication Strategies », dans Retroviruses, Cold Spring Harbor Laboratory Press, (ISBN 0-87969-571-4, lire en ligne)



