Pyroséquençage
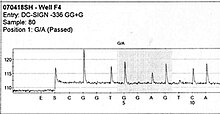
Le pyroséquençage est une technique de séquençage de l'ADN qui permet d’effectuer un séquençage rapide et à moindre coût qu’un séquençage par la méthode de Sanger. En effet, cette technique ne nécessite pas de clonage (donc gain de temps et d’argent), et permet une lecture directe de la séquence obtenue après le séquençage. Elle est basée sur le principe du « séquençage par synthèse », dans lequel le séquençage est effectué en détectant le nucléotide incorporé par une ADN polymérase. Le pyroséquençage s'appuie sur la détection, par quantification de lumière émise, du pyrophosphate libéré lors de la réaction en chaîne, d'où le nom de pyroséquençage.
Histoire
[modifier | modifier le code]Le principe du pyroséquençage a d'abord été énoncé en 1993 par Pettersson, Uhlen et Nyren[1] en combinant la phase solide du séquençage[2] utilisant des billes magnétiques enveloppées de streptavidine avec de l'ADN polymérase recombinante n'ayant pas d'activité 3'-5' exonucléase (permettant la correction sur épreuves ou proof-reading) et de la détection de luminescence via l'activité enzymatique de la luciférase[3]. C'est donc un mélange de trois enzymes (ADN polymérase, ATP sulfurylase et luciférase) et un nucléotide (dNTP) qui est ajouté à un simple brin d'ADN que l'on veut séquencer, et l'incorporation du nucléotide est suivie en mesurant la quantité de lumière émise. L'intensité de la lumière détermine si 0, 1 ou plusieurs nucléotides ont été incorporés, ce qui permet de voir combien de nucléotides complémentaires sont présents sur le brin matrice. La mixture d'enzymes avec le premier nucléotide est ensuite retirée du milieu avant qu'une autre mixture avec le deuxième nucléotide soit ajoutée. Ce processus est répété avec chacun des quatre types de nucléotides jusqu'à ce que la séquence d'ADN du simple brin matrice soit déterminée.
Une seconde méthode de pyroséquençage a été développée en 1998 par Ronaghi, Uhlen et Nyren[4]. On ajoute une enzyme en plus, l'apyrase, qui a pour rôle de dégrader les nucléotides en surplus qui ne sont pas incorporés par l'ADN polymérase. Cela permet à la mixture enzymatique d'être gardée tout au long de la procédure, permettant ainsi au dispositif d'être autonome. Un instrument automatisé basé sur ce principe a été introduit sur le marché l'année suivante par la compagnie Pyrosequencing.
Enfin, une troisième méthode alternative a été développée en 2005 par Rothberg et ses collaborateurs[5]. Elle est basée sur le principe originel d'attacher l'ADN à séquencer sur un support solide et le séquençage se fait de manière similaire aux autres méthodes en utilisant une puce à ADN microfabriquée. Cette méthode a permis un séquençage de l'ADN à haut débit et de développer un instrument automatisé introduit sur le marché par la suite. Il est devenu le premier instrument de séquençage de nouvelle génération, ouvrant ainsi la voie à une nouvelle ère en génomique, avec des coûts diminuant rapidement, permettant un séquençage complet de génomes à des prix abordables.
Procédure[6]
[modifier | modifier le code]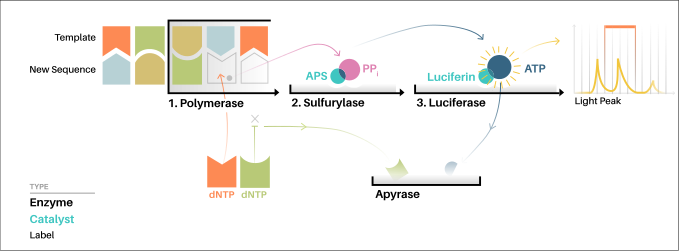
Dans le principe du "séquençage par synthèse", on prend un simple brin d'ADN à séquencer et on synthétise ensuite son brin complémentaire de façon enzymatique.
La méthode de pyroséquençage est basée sur la détection de l'activité de l'ADN polymérase avec une autre enzyme chimioluminescente. Dans son principe, la méthode permet de séquencer un simple brin d'ADN en synthétisant son brin complémentaire à raison d'une paire de bases à la fois, en détectant à chaque fois quelle base a été ajoutée à chaque étape. La matrice d'ADN est immobile et des solutions de nucléotides A, T, G et C sont ajoutées l'une après l'autre puis retirées de la réaction. De la lumière est produite seulement lorsque la solution de nucléotides complémente la première base non appariée de la matrice. La séquence des solutions nucléotidiques produisant des signaux chimioluminescents permet de déterminer la séquence du brin matrice.
En ce qui concerne la méthode développée en 1998, la matrice d'ADN simple brin est hybridée à une amorce et incubée avec les enzymes ADN polymérase, ATP sulfurylase, luciférase et apyrase et avec les substrats 3'-phosphoadénosine 5'-phosphosulfate (PAPS) et luciférine.
- L'ajout d'un des 4 désoxynucléotides triphosphates (le dNTPαS, qui n'est pas un substrat de la luciférase, est ajouté au lieu du dATP afin d'éviter le bruit de fond) permet d'initier la deuxième étape. L'ADN polymérase incorpore les dNTP complémentaires sur la matrice. Cette incorporation libère un pyrophosphate.
- L'ATP sulfurylase convertit le pyrophosphate en ATP en présence de la PAPS. Cet ATP agit comme substrat pour la conversion de la luciférine en oxyluciférine catalysée par la luciférase, ce qui génère de la lumière visible en quantités proportionnelles à la quantité d'ATP. La lumière produite au cours de cette réaction est détectée par une caméra et analysée par un programme informatique : le séquenceur ou plus précisément un capteur CCD va capter ce signal lumineux et le reproduire sous forme d’un pic sur le pyrogramme. La hauteur de ce pic est fonction de l’intensité du signal lumineux, elle-même proportionnelle au nombre de nucléotides incorporés en même temps.
- Les nucléotides non incorporés (en excès) sont dégradés par l'apyrase, et la réaction peut reprendre avec un nouveau nucléotide.
À partir du pyrogramme, on peut donc déduire la séquence à partir de la taille des pics obtenus. Par ailleurs, en cas de mélange de nucléotides à une même position (polymorphisme de séquence), la taille des pics permet d'avoir une quantification de la proportion de brins porteurs de l'un ou l'autre des nucléotides.
Limites
[modifier | modifier le code]Actuellement, l'une des limites de la méthode est que les longueurs des lectures individuelles de la séquence d'ADN sont autour de 300 à 500 nucléotides, soit moins que les 800 à 1000 nucléotides que l'on peut obtenir avec les méthodes à terminaison de chaîne comme la méthode de Sanger. Cela peut rendre le processus d'assemblage du génome plus difficile, en particulier pour les séquences contenant une grande quantité de séquences répétées. Le manque d'activité de correction sur épreuves (proof-reading) limite la précision de cette méthode.
Voir aussi
[modifier | modifier le code]Notes
[modifier | modifier le code]- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Pyrosequencing » (voir la liste des auteurs).
Références
[modifier | modifier le code]- P. Nyren, B. Pettersson et M. Uhlen, « Solid Phase DNA Minisequencing by an Enzymatic Luminometric Inorganic Pyrophosphate Detection Assay », Analytical Biochemistry, vol. 208, no 1, , p. 171–175 (ISSN 0003-2697, DOI 10.1006/abio.1993.1024, lire en ligne, consulté le )
- (en) M. Uhlen, « Magnetic separation of DNA », Nature, vol. 340, no 6236, , p. 733–734 (ISSN 1476-4687, DOI 10.1038/340733a0, lire en ligne, consulté le )
- Pål Nyrén et Arne Lundin, « Enzymatic method for continuous monitoring of inorganic pyrophosphate synthesis », Analytical Biochemistry, vol. 151, no 2, , p. 504–509 (ISSN 0003-2697, DOI 10.1016/0003-2697(85)90211-8, lire en ligne, consulté le )
- (en) Mostafa Ronaghi, Mathias Uhlén et Pål Nyrén, « A Sequencing Method Based on Real-Time Pyrophosphate », Science, vol. 281, no 5375, , p. 363–365 (ISSN 0036-8075 et 1095-9203, PMID 9705713, DOI 10.1126/science.281.5375.363, lire en ligne, consulté le )
- (en) Marcel Margulies, Michael Egholm, William E. Altman et Said Attiya, « Genome sequencing in microfabricated high-density picolitre reactors », Nature, vol. 437, no 7057, , p. 376–380 (ISSN 1476-4687, DOI 10.1038/nature03959, lire en ligne, consulté le )
- « Pyrosequencing Technology and Platform Overview - QIAGEN », sur www.qiagen.com (consulté le )

